|
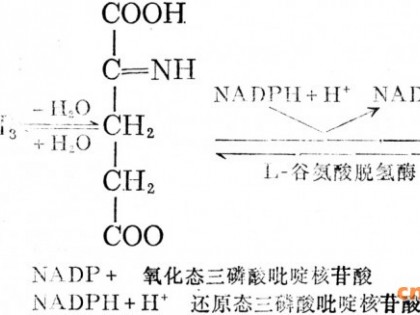
| 图 1 枯草芽孢杆菌L-脯氨酸生物合成途径Figure 1 Biosynthetic pathway of L-proline in Bacillus subtilis. |
|
图选项
|
目前关于枯草芽孢杆菌脯氨酸合成途径的研究基本集中于细胞生理层面[8],包括途径中各基因的表达对胞内游离脯氨酸变化以及自身渗透压调节的影响[2, 5],或者将研究枯草芽孢杆菌与植物的生理条件研究相结合,探究枯草芽孢杆菌作为根系微生物在高渗环境下提升植物生存能力的机制[9-10]。现有的研究已阐明,枯草芽孢杆菌编码谷氨酸激酶(GK)的proB基因在高渗透压胁迫下表达会上调[11],且其表达受到产物L-脯氨酸的反馈抑制[12]。融合表达枯草芽孢杆菌proBA基因能进一步提高菌株的胞内游离脯氨酸含量和极端环境耐受性[13]。而在非胁迫条件下,该途径中各基因对代谢通路的影响未知。
本文从枯草芽孢杆菌以谷氨酸为前体的脯氨酸合成途径出发,分别考察了glnA、proB和proA基因在盐离子胁迫条件下和非胁迫条件下对胞外和胞内游离脯氨酸积累的影响,研究结果有助于相关代谢途径关键节点的鉴定,从而为利用基因工程技术增强微生物及其依附的植物在高渗环境下的耐受性提供了理论依据[13-14]。此外,本研究通过对合成途径的改造,可以有效加强脯氨酸生物合成,进而提高胞外脯氨酸的积累量,为从系统代谢工程水平构建产L-脯氨酸工程菌提供了实验基础[15]。
1 材料和方法1.1 菌株和质粒本研究中所使用的菌株、质粒如表 1所示。
| Stains and plasmids | Relevant characteristics | Sources |
| Strains | ||
| JM109 | E. coli, wild type | This Lab |
| WB600 | Bacillus subtilis, wild type | This Lab |
| WB601 | WB600 harboring pHY-P43-proB plasmid | This study |
| WB602 | WB600 harboring pHY-P43-proA plasmid | This study |
| WB603 | WB600(ΔglnA, glnA∷Neo), Neor | This study |
| WB604 | WB600[ΔglnA, glnA∷(P43-proB-Neo)], Neor | This study |
| Plasmids | ||
| pMA5 | Bacillus-E. coli shuttle vector, Ampr and Neor | This Lab |
| pMD19-T | Cloning vector, Ampr | This Lab |
| pMD19T-glnA | pMD19-T based vector, carrying glnA gene, Ampr | This study |
| pMD19T-Neo | pMD19-T based vector, carrying Neo gene, Ampr | This study |
| pMD19T-ΔglnA∷Neo | pMD19-glnA based vector, Amprand Neor | This study |
| pMD19T-P43-proB | pMD19-T based vector, Ampr | This study |
| pMD19T-ΔglnA∷P43-proB-Neo | pMD19T-ΔglnA∷Neo based vector, Ampr and Neor | This study |
| pHY-P43 | Bacillus-E. coli shuttle vector, Amprand Tetr | This Lab |
| PHY-P43-proB | pHY-P43 based vector, carrying proB gene, Ampr and Tetr | This study |
| pHY-P43-proA | pHY-P43 based vector, carrying proA gene, Ampr and Tetr | This study |
1.2 工具酶与试剂
本研究所用的Taq DNA聚合酶、Pfu DNA聚合酶、T4连接酶均购于大连宝生物工程有限公司;pMD19-T vector购自TaKaRa公司;试剂新霉素、氨苄青霉素、四环素购自Sigma公司;质粒DNA抽提试剂盒、DNA纯化试剂盒和DNA凝胶回收试剂盒购于北京博大泰克生物有限公司。蛋白胨、酵母粉、琼脂粉购于OXOID公司;其他试剂均为国产或进口分析纯。
1.3 培养基与培养条件LB培养基(g/L):蛋白胨10,酵母粉5,NaCl 10,固体LB培养基需加入1.5%的琼脂粉。TB发酵培养基(g/L):蛋白胨12.00,酵母粉24.00,K2HPO4 12.54,KH2PO4 2.30,pH 7.0。摇瓶发酵装液量为30 mL/250 mL。在菌株构建和发酵过程中,培养基中添加的氨苄青霉素、新霉素、四环素终浓度分别为100、6、20 mg/L。
1.4 引物本研究克隆新霉素基因Neo所用的引物是根据pMA5质粒上新霉素抗性基因序列设计而成,其余所用引物根据GenBank中Bacillus subtilis 168的glnA、proB、proA和P43的基因序列信息设计而成,本实验所用引物的合成及相关测序均由苏州金唯智生物科技有限公司完成,具体序列见表 2。
| Primers | Sequences (5′→3′) |
| glnA-FW | ATGGCAAAGTACACTAGAGAAG |
| glnA-RV | TTAATACTGAGACATATACTGT |
| NEO-FW | ACGCCATGGAGGATTGAAGGATGCT |
| NEO-RV | CCACTGCAGAGCACACCCTTTATTC |
| P43N-FW | CATGCCATGGTGTCGACGTGCATGCAGGCC |
| P43N-RV | CATGCCATGGTATAATGGTACCGCTATCACTTT |
| proBB-FW | CGCGGATCCATGAAAAAACAAAGAATAG |
| proBE-RV | CCGGAATTCCTAGTCTTTTACATTGAC |
| proBN-RV | CATGCCATGGCTAGTCTTTTACATTGACCC |
| proA-FW | CGGGATCCATGAGTGAAG TTTCTGTAAA A |
| proA-RV | TCCCCCGGGCTATTCACGG ATTTGCCCAGTT |
| LJ-P43B | TATTCTTTGTTTTTTCATTATAATGGTACCGCTATCAC |
| LJ-BP43 | TATAAAGTGATAGCGGTACCATTATAATGAAAAAACAAAGAATAGT |
| Underline sequences are cleavage site of restriction enzyme. | |
1.5 重组质粒的构建
利用引物proBB-FW & proBE-RV从枯草芽孢杆菌WB600基因组上将编码谷氨酸激酶的proB基因克隆出来,连接到经相同酶切的pHY-P43载体上,构建质粒pHY-P43-proB。利用类似方法构建了重组质粒pHY-P43-proA。
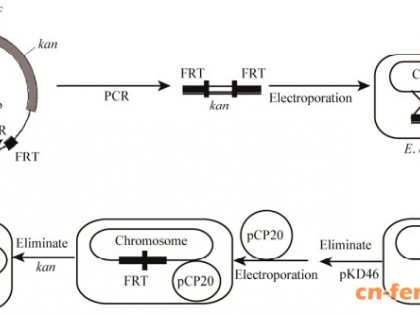
1.6 枯草芽孢杆菌目的基因的敲除枯草芽孢杆菌目的基因的敲除采用同源重组的方法[16]。本实验首先利用谷氨酰胺合成酶基因glnA上下游引物glnA-FW和glnA-RV扩增出glnA基因序列,大小为1335 bp。然后将其连接到pMD19-T质粒上,构建质粒pMD19T-glnA。同样的,利用引物NEO-FW和NEO-RV扩增出新霉素抗性基因Neo,并将其连接到pMD19-T载体上,构建质粒pMD19T-Neo。将质粒pMD19T-glnA和pMD19T-Neo均用Nco Ⅰ和Pst Ⅰ酶进行双酶切,酶切后的新霉素片段连接到经同样酶切的pMD19T-glnA质粒上,构建敲除质粒pMD19T-ΔglnA∷Neo。最后将敲除质粒pMD19T-ΔglnA∷Neo转化进入枯草芽孢杆菌WB600中进行同源重组,在新霉素平板上筛选正确的转化子。敲除质粒的构建如图 2所示。
 |
| 图 2 敲除质粒pMD19T-ΔglnA∷Neo和pMD19T-ΔglnA∷P43-proB-Neo的构建Figure 2 Cloning strategy of knockout plasmids pMD19T-ΔglnA∷Neo and pMD19T-ΔglnA∷P43-proB-Neo. |
|
图选项
|
1.7 重叠片段P43-proB的整合
首先利用引物P43N-FW和P43N-RV,proBB-FW和proBN-RV分别从枯草芽孢杆菌WB600的基因组上扩增出启动子P43以及谷氨酸激酶基因proB的序列。然后利用引物LJ-P43B和LJ-BP43将二者通过重叠PCR连接成片段P43-proB,并将此片段经Nco Ⅰ单酶切后连接到同样经Nco Ⅰ单酶切的敲除质粒pMD19T-ΔglnA∷Neo上,构建了重组质粒pMD19T-ΔglnA∷P43-proB-Neo。然后将此重组质粒转化进入枯草芽孢杆菌WB600中进行同源重组,在新霉素抗性平板上筛选正确的转化子。具体的构建方法见图 2。
1.8 重组菌生长曲线的绘制将重组菌接种于20 mL LB液体培养基中,37℃、200 r/min过夜活化。按照初始OD600 0.1的添加量转接于30 mL LB液体培养基中,37℃、200 r/min培养36 h,期间每隔2 h取样1次测定菌体浓度,最后一次取样间隔为4 h。菌体浓度的测定以分光光度计600 nm波长下的吸光值OD600表示。OD600根据下列公式转化为细胞干重[17]:1 OD =0.35 g/L DCW。
1.9 重组菌摇瓶发酵将重组菌接种于20 mL LB液体培养基中,37℃、200 r/min活化12 h。按初始OD600 0.1的接种量转接至50 mL的TB培养基中,菌株WB603和WB604初始培养基中添加终浓度为5 mmol/L的谷氨酰胺,37℃、250 r/min摇瓶发酵24 h。发酵结束时取样测量OD600以及发酵液中L-脯氨酸含量,并收集细胞测定胞内游离脯氨酸含量。
1.10 L-脯氨酸的测定L-脯氨酸的测定采用酸性茚三酮法[2, 18]。胞外脯氨酸的测定:发酵液12000 r/min离心30 min后,取上清加入等体积10%三氯乙酸沉淀4 h,12000 r/min离心20 min后取上清,稀释适当倍数后测定胞外脯氨酸含量。胞内游离脯氨酸测定:收集菌体,菌体用去离子水洗涤2次,重悬菌体并调整到OD600值为5,加入终浓度为20 mg/L的溶菌酶37℃保温3 h后参照Whatmore AM的方法[2]测定。
2 结果和讨论2.1 含重组质粒枯草芽孢杆菌的构建按照1.5节的方法构建了质粒pHY-P43-proB和pHY-P43-proA,并转化入大肠杆菌JM109中,转化子提取质粒酶切验证。其中,质粒pHY-P43-proB经BamH Ⅰ & EcoR Ⅰ双酶切后片段大小分别为5278 bp、1098 bp;质粒pHY-P43-proA经BamH Ⅰ & SmaⅠ双酶切后片段大小分别为5278 bp和1248 bp,此结果表明重组质粒构建成功,如图 3。分别将二者转化入枯草芽孢杆菌WB600原始菌株中,构建了菌株WB601和WB602。
 |
| 图 3 重组质粒示意图Figure 3 Recombinant plasmids map. |
|
图选项
|
2.2 glnA基因缺失重组菌的构建
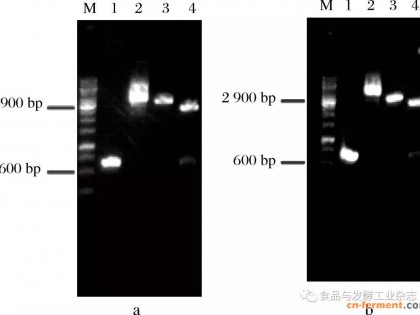
按照上述1.6节的方法构建了敲除质粒pMD19T-ΔglnA∷Neo,并将其转化入WB600原始菌株中,转化子以glnA-FW和Neo-RV为引物经菌落PCR后得到大小为1241 bp的片段。结果表明成功敲除了谷氨酰胺合成酶基因glnA,即构建了菌株WB603。
2.3 P43-proB基因片段整合到基因组glnA位点重组菌的构建按照1.7节的方法将启动子P43与proB基因连接成P43-proB基因片段,大小为1524 bp。同时,在1.6构建的敲除盒基础上,将P43-proB片段连接至pMD19T-ΔglnA∷Neo质粒上,构建敲除质粒pMD19T-ΔglnA∷P43-proB-Neo,并将其转化入原始菌WB600中。转化子以glnA-FW和glnA-RV为引物菌落PCR后得到大小为3051 bp的片段;以glnA-FW和proB-RV为引物菌落PCR后得到大小为1765 bp的片段,结果表明P43-proB片段成功整合到原始菌株基因组glnA位点上,即构建了重组菌WB604。
2.4 glnA基因缺失以及P43-proB片段整合到基因组上对重组菌生长的影响为判断谷氨酰胺合成酶基因glnA的缺失对重组菌生长的影响,将重组菌WB603和WB604按1.8节所示方法接种于LB培养基中,绘制其菌体生长曲线。重组菌WB603生长状况如图 4-A所示,在不添加谷氨酰胺的情况下生长缓慢,最大OD600仅能达到4左右,约为原始菌的60%;而在添加了5 mmol/L谷氨酰胺的情况下,WB603生长加快,延滞期明显缩短,最大OD600可以达到5.7左右,但仍与原始菌存在差距。此结果表明,glnA基因的缺失在改变重组菌基因组结构的同时,也影响了菌体正常生理代谢,进而使其无法恢复正常生长。因此,glnA基因的缺失会形成谷氨酰胺部分营养缺陷型,而通过外源添加谷氨酰胺可部分回补细胞的营养型生长[19]。
 |
| 图 4 重组菌WB603和WB604生长曲线Figure 4 Growth curves of recombinant strains WB603 and WB604. +Gln means initial LB medium contained 5 mmol/L glutamine. |
|
图选项
|
重组菌WB604生长与WB603类似,在培养基中添加终浓度5 mmol/L谷氨酰胺时,其生长状况明显优于未添加谷氨酰胺,如图 4-B所示。
2.5 非胁迫条件下重组菌胞外和胞内游离脯氨酸积累情况为考察不同重组菌株WB601、WB602、WB603、WB604合成L-脯氨酸的能力,分别对这4株菌进行摇瓶发酵,对照组为原始菌株WB600。发酵条件:TB培养基37℃、250 r/min摇瓶培养24 h,结果如表 3所示。
| Strain | OD600 | Extracellular L-proline/ (mg/L) | Extracellular L-proline yield/ (mg/g DCW) | Intracellular free L-proline yield/ (mg/g DCW) |
| WB600 | 16.34±0.03 | 194.53±1.41 | 34.36±4.54 | 5.06±1.24 |
| WB600 (+Gln) | 15.95±0.07 | 215.87±6.19 | 39.57±5.21 | 6.49±2.80 |
| WB601 | 8.74±0.08 | 430.31±9.24 | 140.61±4.24 | 9.66±2.07 |
| WB602 | 4.68±0.03 | 548.28±7.69 | 336.89±11.62 | 16.90±2.04 |
| WB603 | 11.17±0.05 | 1221.43±10.06 | 313.56±8.84 | 18.53±0.95 |
| WB603 (+Gln) | 13.17±0.10 | 925.27±9.56 | 201.23±7.43 | 25.52±1.81 |
| WB604 | 11.20±0.05 | 1391.65±11.54 | 357.78±9.67 | 24.57±2.53 |
| WB604 (+Gln) | 13.65±0.78 | 992.48±8.79 | 208.59±6.35 | 26.90±1.52 |
| +Gln means initial medium contained 5 mmol/L glutamine. | ||||
在无胁迫条件下,过表达proB和proA的重组菌WB601和WB602其胞外脯氨酸含量分别是原始菌的2.21和2.82倍,单位细胞胞外脯氨酸得率分别是原始菌的4.09倍和9.80倍,而胞内游离脯氨酸含量分别是原始菌的1.91倍和3.34倍。此结果表明,proB和proA基因的过表达能够显著增强细胞合成脯氨酸的能力,这是因为proB和proA基因的过表达增加了脯氨酸合成途径的代谢通量,从而提高了脯氨酸的积累量。然而,proB和proA基因的过表达也会对菌体生长带来负担(最大菌体量仅为原始菌的53%和28%左右),其中proA基因的过表达对生长的负面作用更显著。在枯草芽孢杆菌中,proB和proA基因分别编码谷氨酸激酶和谷氨酰磷酸还原酶,过表达proB和proA时一方面使得细胞将更多的能量用于酶的表达;另一方面,基因的过表达可能使得野生菌细胞内平衡的代谢流发生了扰动[20],从而影响了生长。此外,proB基因编码的谷氨酸激酶过表达时会使得L-脯氨酸积累更多[21],过量的脯氨酸会对谷氨酸激酶造成一定程度的反馈抑制[7],而proA基因编码的谷氨酰磷酸还原酶则不会出现这种情况,从表中也可知过表达proA基因相比过表达proB基因更有利于脯氨酸的合成。因此,谷氨酰磷酸还原酶的表达效果可能要优于谷氨酸激酶的表达,因而过表达proA基因对生长造成的负担大于过表达proB基因[22]。
在不添加谷氨酰胺时,glnA基因缺失的重组菌WB603胞外脯氨酸含量为1221.43 mg/L,是原始菌的6.28倍,单位细胞胞外和胞内游离脯氨酸分别为原始菌的9.13倍和3.66倍。这表明,glnA基因的缺失提高了细胞合成脯氨酸的能力,并且对胞外脯氨酸的合成影响更显著。由此推断,谷氨酰胺合成酶基因glnA的敲除阻断了谷氨酸的竞争支路,增加了脯氨酸从头合成途径中的前体物质谷氨酸含量,结果使代谢流更多向脯氨酸积累,从而加强了脯氨酸的生物合成。此外,本研究中glnA基因的缺失使得重组菌在积累胞外脯氨酸方面要优于proB和proA基因的过表达,这可能是因为细胞内基因的敲除与过表达对细胞造成的影响不同,从而造成了细胞合成代谢物质的能力有所差异。而在本实验中,glnA基因的缺失对生长造成的影响小于proB和proA基因的过表达,从而可能造成了其胞外脯氨酸的积累高于后二者。与此同时,在相同条件下,重组菌WB604的菌体量与WB603几乎相同,表明在基因组glnA位点上插入P43-proB片段与单独敲除glnA基因相比对菌体生长几乎没有影响。但重组菌WB604胞外积累了1391.65 mg/L脯氨酸,单位细胞得率为357.78 mg/g DCW,相比菌株WB603,胞外脯氨酸含量提高了13.94%,单位细胞胞外脯氨酸得率提高了14.10%,且胞内游离脯氨酸含量提高了32.60%。这表明,对脯氨酸合成途径关键节点glnA和proB的同时改造可使正向协同效应叠加,进一步提升了细胞合成脯氨酸的能力。
另一方面,不添加Gln (谷氨酰胺)时重组菌生长变差,但较添加后相比区别不大。此外,对于胞外脯氨酸来说,不添加Gln时重组菌WB603和WB604胞外脯氨酸含量分别为1221.43 mg/L和1391.65 mg/L,分别是添加Gln时的1.32倍和1.40倍。值得注意的是,添加Gln时重组菌WB603和WB604的胞内游离脯氨酸含量均有所提高,分别为不添加时的1.38倍和1.09倍,而Gln的加入对原始菌胞外和胞内游离脯氨酸含量影响不大。此结果表明,Gln的加入能够部分弥补重组菌因基因缺失而造成的生长缺陷,然而Gln的加入在促进重组菌生长的同时,也可能会促使菌株利用胞外脯氨酸作为碳源或氮源来合成其他代谢物质[23],从而造成胞外脯氨酸含量有所降低,胞内游离脯氨酸浓度有所提高[24]。
2.6 盐离子胁迫条件下重组菌胞外和胞内游离脯氨酸积累情况为探究重组菌在盐离子胁迫下脯氨酸的积累情况,在2.5节重组菌培养的基础上,在培养基中添加5%的NaCl,其余条件不变,摇瓶培养24 h后测定重组菌胞外和胞内游离脯氨酸含量,测定结果见表 4。
| Strains | OD600 | Extracellular L-proline/ (mg/L) | Extracellular L-proline yield/ (mg/g DCW) | Intracellular free L-proline yield/ (mg/g DCW) |
| WB600 | 13.47±0.24 | 248.44±5.25 | 52.37±8.49 | 51.10±1.10 |
| WB600 (+Gln) | 13.79±0.02 | 296.32±7.99 | 64.21±6.06 | 50.98±1.93 |
| WB601 | 12.25±0.04 | 482.67±8.15 | 112.46±4.04 | 50.59±1.29 |
| WB602 | 9.61±0.04 | 381.51±4.24 | 114.79±6.36 | 50.67±0.55 |
| WB603 | 7.74±0.09 | 1032.74±11.88 | 381.68±8.15 | 49.65±2.98 |
| WB603 (+Gln) | 10.12±0.06 | 728.42±8.49 | 206.93±3.54 | 47.75±1.59 |
| WB604 | 9.73±0.02 | 1369.54±13.69 | 402.84±9.26 | 54.88±0.95 |
| WB604 (+Gln) | 11.08±0.57 | 783.91±5.66 | 202.59±4.49 | 52.62±1.69 |
| +Gln means initial medium contained 5 mmol/L glutamine. | ||||
在含5% NaCl的培养基中,实验组菌株生长均受到不同程度的胁迫,而胞内游离脯氨酸含量均高于非胁迫条件下,这与所报道的在盐离子胁迫条件下细胞内的游离脯氨酸浓度会有所提高一致[5],并且实验组菌株的胞内游离脯氨酸浓度差异不大,这可能与胞内游离脯氨酸平衡有关[2]。值得一提的是,过表达proB和proA基因的重组菌WB601和WB602相比于非胁迫条件下,菌体量却有所提高(可参比非胁迫条件下的表 3)。此外,在glnA基因缺失的基础上进一步过表达proB基因的重组菌WB604在盐离子胁迫条件下的生长要优于重组菌WB603。由此可以得出结论,proB和proA基因的过表达能够在一定程度上增强菌株的耐盐性。
对重组菌WB601和WB602的胞外脯氨酸积累情况分析可得,其胞外脯氨酸含量分别是原始菌的1.94倍和1.54倍,单位细胞胞外脯氨酸得率分别是原始菌的2.15倍和2.19倍,差别不大且较非胁迫时相比有所降低。与此同时,在不添加Gln时,glnA基因缺失的重组菌WB603胞外脯氨酸含量及单位细胞得率分别是原始菌的4.16倍和7.29倍,同样低于非胁迫条件下。相同条件下,相比于WB603,重组菌WB604的胞外脯氨酸含量及其单位细胞得率分别提高了32.61%和5.54%,较非胁迫时相比胞外脯氨酸提升幅度增加,而单位细胞胞外脯氨酸得率有所降低,这可能与在盐离子胁迫时过表达proB基因的重组菌WB604菌体生长受到的影响较WB603小有关。以上结果表明,在盐离子胁迫条件下,glnA基因的缺失同样能提高细胞合成脯氨酸的能力;而在此基础之上过表达proB基因进一步提高了细胞合成脯氨酸的能力,但受盐离子胁迫的干扰。此外,在添加Gln时,重组菌WB603和WB604的胞外脯氨酸含量分别比不添加时降低了29.47%和42.76%,且单位细胞胞外脯氨酸得率也明显降低。这可能因为是在盐离子胁迫条件下,菌株为了应对胁迫环境,一方面通过自身的保护机制吸收外界脯氨酸维持体内渗透压[4],一方面可能利用胞外脯氨酸来尽可能合成生长需要的营养物质[23],而生长有缺陷的基因工程菌对此需求更大。值得注意的是,原始菌胞外脯氨酸含量较非胁迫时相比均有所提高,这可能是胞内游离脯氨酸达到平衡后溢出所致[2]。
3 结论本研究以枯草芽孢杆菌谷氨酸到脯氨酸代谢途径中glnA、proB、proA基因为研究对象,通过分子改造分别构建了重组菌WB601-WB604,摇瓶发酵检测各重组菌的胞外和胞内游离脯氨酸积累情况,以此探究途径中相关基因的功能以及对渗透压调节因子脯氨酸合成的影响。结果显示,在非胁迫条件下,proB、proA基因的过表达能显著提高细胞合成脯氨酸的能力,说明proB、proA基因的过表达增强了脯氨酸合成途径。但是过表达proB、proA会对菌体生长造成负担,而过表达proA对生长的影响更大,进而可能影响了其胞外脯氨酸的积累;glnA基因的缺失同样能增强细胞合成脯氨酸的能力,表明敲除glnA基因阻断了脯氨酸代谢途径中前体谷氨酸的分解途径,从而加强了脯氨酸合成途径。此外,相比于单独敲除glnA基因,在glnA基因缺失的基础上过表达proB基因使得重组菌脯氨酸的合成能力进一步提升。这表明,以上2个关键节点的同时改造可对细胞合成脯氨酸的能力产生正向协同作用。
在盐离子胁迫条件下,实验菌株生长均受到不同程度的胁迫,而胞内游离脯氨酸含量均高于非胁迫条件下,并达到相对平衡的状态,这说明在盐离子胁迫条件下,细胞内的游离脯氨酸浓度会上升并达到相对平衡状态从而应对高渗环境。此外,重组菌WB601、WB602和WB604的菌体量均高于非胁迫时,说明过表达proB、proA基因有助于提高细胞的耐盐性。与此同时,与非胁迫条件下相同,过表达proB、proA基因以及glnA基因的缺失均能显著增强细胞合成胞外脯氨酸的能力,在glnA基因缺失的基础上过表达proB基因使得重组菌脯氨酸的合成能力进一步提高。但其胞外脯氨酸含量均较非胁迫时有所下降,这可能与胁迫条件下菌株利用胞外脯氨酸来维持自身生长代谢以应对高渗环境有关[3, 23]。
| [1] | Schallmey M, Singh A, Ward OP. Developments in the use of Bacillus species for industrial production. Canadian Journal of Microbiology, 2004, 50(1): 1-17. DOI:10.1139/w03-076 |
| [2] | Whatmore AM, Chudek JA, Reed RH. The effects of osmotic upshock on the intracellular solute pools of Bacillus subtilis. Journal of General Microbiology, 1990, 136(12): 2527-2535. |
| [3] | Kempf B, Bremer E. Uptake and synthesis of compatible solutes as microbial stress responses to high-osmolality environments. Archives of Microbiology, 1998, 170(5): 319-330. DOI:10.1007/s002030050649 |
| [4] | von Blohn C, Kempf B, Kappes RM, Bremer E. Osmostress response in Bacillus subtilis:Characterization of a proline uptake system (OpuE) regulated by high osmolarity and the alternative transcription factor sigma B. Molecular Microbiology, 1997, 25(1): 175-187. DOI:10.1046/j.1365-2958.1997.4441809.x |
| [5] | Brill J, Hoffmann T, Bleisteiner M, Bremer E. Osmotically controlled synthesis of the compatible solute proline is critical for cellular defense of Bacillus subtilis against high osmolarity. Journal of Bacteriology, 2011, 193(19): 5335-5346. DOI:10.1128/JB.05490-11 |
| [6] | Belitsky BR, Brill J, Bremer E, Sonenshein AL. Multiple genes for the last step of proline biosynthesis in Bacillus subtilis. Journal of Bacteriology, 2001, 183(14): 4389-4392. DOI:10.1128/JB.183.14.4389-4392.2001 |
| [7] | Brill J, Hoffmann T, Putzer H, Bremer E. T-box-mediated control of the anabolic proline biosynthetic genes of Bacillus subtilis. Microbiology, 2011, 157: 977-987. DOI:10.1099/mic.0.047357-0 |
| [8] | Hayat S, Hayat Q, Alyemeni MN, Wani AS, Pichtel J, Ahmad A. Role of proline under changing environments:a review. Plant Signaling & Behavior, 2012, 7(11): 1456-1466. |
| [9] | Marulanda A, Barea JM, Azcón R. Stimulation of plant growth and drought tolerance by native microorganisms (AM fungi and bacteria) from dry environments:mechanisms related to bacterial effectiveness. Journal of Plant Growth Regulation, 2009, 28(2): 115-124. |
| [10] | Vardharajula S, Ali SZ, Grover M, Reddy G, Bandi V. Drought-tolerant plant growth promoting Bacillus spp.:effect on growth, osmolytes, and antioxidant status of maize under drought stress. Journal of Plant Interactions, 2011, 6(1): 1-14. |
| [11] | Kunst F, Rapoport G. Salt stress is an environmental signal affecting degradative enzyme synthesis in Bacillus subtilis. Journal of Bacteriology, 1995, 177(9): 2403-2407. DOI:10.1128/jb.177.9.2403-2407.1995 |
| [12] | Chen MQ, Cao JW, Zheng CY, Liu Q. Directed evolution of an artificial bifunctional enzyme, γ-glutamyl kinase/γ-glutamyl phosphate reductase, for improved osmotic tolerance of Escherichia coli transformants. FEMS Microbiology Letters, 2006, 263(1): 41-47. DOI:10.1111/fml.2006.263.issue-1 |
| [13] | Chen MQ, Wei HB, Cao JW, Liu RJ, Wang YL, Zheng CY. expression of Bacillus subtilis proBA genes and reduction of feedback inhibition of proline synthesis increases proline production and confers osmotolerance in Transgenic Arabidopsis. BMB Reports, 2007, 40(3): 396-403. DOI:10.5483/BMBRep.2007.40.3.396 |
| [14] | Liu RJ, Chen T, Cao JW. Construction of fused osmoregulation proBA gene from a salt-tolerant mutant of Bacillus subtilis and its influence on the osmotolerance of Escherichia coli. Acta Microbiologica Sinica, 2005, 45(1): 23-26. (in Chinese) 刘瑞杰, 陈婷, 曹军卫. 渗透压调节基因proBA的融合表达对大肠杆菌耐高渗胁迫能力的影响. 微生物学报, 2005, 45(1): 23-26. |
| [15] | Park JH, Lee SY. Towards systems metabolic engineering of microorganisms for amino acid production. Current Opinion in Biotechnology, 2008, 19(5): 454-460. DOI:10.1016/j.copbio.2008.08.007 |
| [16] | Peng QA, Zhang XF, Wu SF, Wang WP. Construction of Bacillus subtilis tkt-mutant by homologous recombination. Biotechnology, 2006, 16(6): 23-26. (in Chinese) 彭其安, 张西峰, 吴思方, 王伟平. 同源重组法构建枯草芽孢杆菌转酮酶缺失突变菌株. 生物技术, 2006, 16(6): 23-26. |
| [17] | 刘延峰. 代谢工程改造枯草芽孢杆菌高效合成N-乙酰氨基葡萄糖. 江南大学博士学位论文, 2015. |
| [18] | Zhi MX, Li XJ. Improvement on the method for measuring proline content. Plant Physiology Communications, 2005, 41(3): 355-357. (in Chinese) 职明星, 李秀菊. 脯氨酸测定方法的改进. 植物生理学通讯, 2005, 41(3): 355-357. |
| [19] | Schreier HJ, Sonenshein AL. Altered regulation of the glnA gene in glutamine synthetase mutants of Bacillus subtilis. Journal of Bacteriology, 1986, 167(1): 35-43. DOI:10.1128/jb.167.1.35-43.1986 |
| [20] | Varma A, Palsson BO. metabolic flux balancing:basic concepts, scientific and practical use. Nature Biotechnology, 1994, 12(10): 994-998. DOI:10.1038/nbt1094-994 |
| [21] | Pérez-Arellano I, Carmona-Álvarez F, Gallego J, Cervera J. Molecular mechanisms modulating glutamate kinase activity. Identification of the proline feedback inhibitor binding site. Journal of Molecular Biology, 2010, 404(5): 890-901. DOI:10.1016/j.jmb.2010.10.019 |
| [22] | Seo JH, Bailey JE. Effects of recombinant plasmid content on growth properties and cloned gene product formation in Escherichia coli. Biotechnology and Bioengineering, 1985, 27(12): 1668-1674.DOI:10.1002/(ISSN)1097-0290 |
| [23] | Moses S, Sinner T, Zaprasis A, Stöveken N, Hoffmann T, Belitsky BR, Sonenshein AL, Bremer E. Proline utilization by Bacillus subtilis:uptake and catabolism. Journal of Bacteriology, 2012, 194(4): 745-758.DOI:10.1128/JB.06380-11 |
| [24] | Zaprasis A, Bleisteiner M, Kerres A, Hoffmann T, Bremer E. Uptake of amino acids and their metabolic conversion into the compatible solute proline confers osmoprotection to Bacillus subtilis. Applied and Environmental Microbiology, 2015, 81(1): 250-259. DOI:10.1128/AEM.02797-14 |