西南大学药学院, 现代生物医药研究所, 重庆 400716 收稿日期: 2018-01-24; 修回日期: 2018-03-26 基金项目: 国家自然科学基金资助项目(31670050,31100069);中央高校基本科研业务费专项资金资助项目(XDJK2017B011)*通讯作者: 廖国建, Tel/Fax: 86-23-68251225, E-mail: gjliao@swu.edu.cn
摘要: 作为首个进入临床应用的新型环脂肽抗生素,达托霉素抗菌活性强,耐药比例低,已成为治疗耐药革兰阳性菌所致感染的一线用药。本文主要对达托霉素的抗菌谱、生物合成途径、作用机制、耐药机制、构效关系、新型衍生物surotomycin和铁载体-达托霉素偶联物杀灭耐药鲍曼不动杆菌等研究结果进行综述,以期为新型达托霉素衍生物研发有所裨益。
关键词: 脂肽 达托霉素 抗菌谱 作用机制 耐药机制 构效关系
Current status and future perspectives of daptomycin development
PENG Xi-xi, WANG Sai-sai, FU Chen, HU Chang-hua, LIAO Guo-jian
Institute of Modern Biopharmaceuticals, School of Pharmaceutical Sciences, Southwest University, Chongqing 400716, China
Abstract: Due to the potent bactericidal activity and low incidence of drug resistance, the novel cyclic lipopeptide antibiotic - daptomycin has emerged as one of the first line antimicrobial agents in the treatment of serious infections caused by gram-positive resistant pathogens. This review summarizes the research advances of daptomycin in recent years, mainly including spectrum of antimicrobial activity, biosynthesis, mode of action, mechanism of drug resistance, structure-activity relationships, surotomycin and siderophore-daptomycin conjugate to kill multidrug resistant Acinetobacter baumannii. The findings summarized in this review highlight the directions of next-generation of daptomycin derivatives.
Key words: lipopeptide daptomycin spectrum of antimicrobial activity mode of action mechanism of drug resistance structure-activity relationship
达托霉素是由玫瑰孢链霉菌(Streptomyces roseosporus)产生的一种新型环脂肽抗生素, 包含1个由10个氨基酸残基组成的大环核心和3个环外氨基酸连接1个癸酸脂肪酸侧链(图 1A)。达托霉素以钙离子依赖的方式与细胞膜相互作用并发挥杀菌活性。带正电的达托霉素-钙离子复合物主要与细胞膜中带负电荷的磷脂酰甘油(phosphatidylglycerol, PG)和心磷脂(cardiolipin, CL)等相互作用, 造成细胞膜去极化, 钾离子等细胞内容物外排, 最终导致细菌死亡[1]。达托霉素在体内外对耐药革兰阳性致病菌具有强力杀菌作用。美国食品药品管理局分别于2003年和2006年批准了默沙东公司研发的达托霉素用于治疗革兰阳性菌引起的复杂皮肤感染和结构性皮肤感染, 以及金黄色葡萄球菌导致的菌血症和右侧心内膜炎[2]。2016年, 我国食品药品监督管理总局批准了华东医药和恒瑞医药生产达托霉素。此外, 达托霉素也被用于治疗由耐万古霉素肠球菌(vancomycin-resistant Enterococcus, VRE)感染引起的脑膜炎、心内膜炎和尿路感染等。由于达托霉素的大量使用, 在金黄色葡萄球菌、肠球菌等主要病原菌中已发现耐药性。然而, 多项研究表明达托霉素上市10多年来耐药比例并未显著提高, 彰显出达托霉素独特的优势[3]。

Figure 1 Figure 1 Chemical structure of daptomycin (A) and its biosynthetic gene cluster (B)
由于日益重要的临床价值, 达托霉素的生物合成机制、构效关系、新型衍生物的合成、达托霉素作用和耐药机制都是非常活跃的研究领域, 取得了重要的进展。本文总结这些领域的最新进展, 特别是构效关系研究以及艰难梭菌感染(Clostridium difficile infection, CDI)治疗的新选择的达托霉素衍生物surotomycin。
1 达托霉素抗菌谱
达托霉素抗菌谱广, 对VRE、耐甲氧西林金黄色葡萄球菌(methicillin-resistant Staphylococcus aureus, MRSA)和耐青霉素的链球菌在体外都有快速杀菌作用。达托霉素体外在1 h内能杀死99.9% MRSA和甲氧西林敏感金黄色葡萄球菌(methicillin-sensitive S. aureus, MSSA), 在心内膜炎模型中, 达托霉素对高密度(1×109 cfu)处于稳定期的MSSA和MRSA仍保持杀菌活性(24 h杀灭99.9%)[4]。虽然美国食品药品管理局未批准达托霉素用于治疗VRE感染, 但是该抗生素已经成为治疗严重VRE感染的一线用药。体外研究发现, 达托霉素对肺炎链球菌有很强的杀菌活性, 但是由于肺部气道具有独特的组织构造, 含有由蛋白和脂类混合物构成的表面活性剂, 影响了抗生素的活性, 因此达托霉素尚不能用于治疗肺炎链球菌引起的肺炎[5]。
2 达托霉素的生物合成
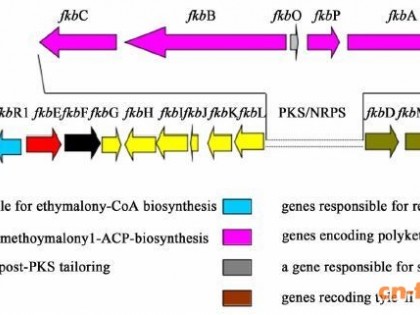
达托霉素是通过非核糖体肽合成酶(non-ribosomal peptide synthetase, NRPS)合成的(图 1B)。生物合成基因簇中的dptA、dptBC和dptD分别编码NRPS的3个亚基DptA、DptBC和DptD。dptE和dptF分别编码酰基-辅酶A连接酶(DptE)和酰基载体蛋白(DptF), 这两个蛋白负责脂肪酸活化, 是达托霉素的起始合成模块。DptA负责催化Trp1、D-Asn2、Asp3、Thr4和Gly5的缩合。DptBC负责催化Orn6、Asp7、D-Ala8、Asp9、Gly10和D-Ser11的缩合。mGlu12和Kyn13由DptD催化合成。整条肽链合成后, Thr4的羟基作为亲核基团攻击Kyn13的羧基形成酯键, 最终形成1个由10个氨基酸残基组成的肽环和3个氨基酸残基组成的肽尾[6]。基因簇还包括可能参与抗性、转运、修饰和调控等基因。dptI编码甲级转移酶, 催化mGlu12的合成, dptJ编码色氨酸2, 3-双加氧酶, 参与Kyn13的形成。dptG编码MbtH同源蛋白, 可能参与氨基酸的修饰, dptH可能参与了达托霉素生物合成过程中的纠错, dptP编码DedA家族蛋白, 可能参与达托霉素抗性, dptM/dptN编码ABC转运蛋白, 其功能未知[7]。dptR1、dptR2和dptR3编码的转录因子通过间接方式调控达托霉素生物合成[8, 9]。
3 达托霉素的作用机制
达托霉素通过钙离子(Ca2+)依赖的方式靶向革兰阳性菌细胞膜。最新的研究表明在枯草芽孢杆菌中, 达托霉素优先插入到富含液脂的位置, 造成细胞膜双分子层的扭曲, 导致液脂聚集到插入位点补偿细胞膜流动性受到的影响。催化细胞壁和细胞膜磷脂合成的关键酶定位于细胞膜中富含液脂的位置, 如负责催化脂质Ⅱ (lipid Ⅱ)最后一步合成的MurG, 催化磷脂合成的PlsX等。达托霉素阻碍了MurG和PlsX等重要蛋白与液脂的结合, 导致蛋白定位异常和功能受损[10]。
通过干扰细胞膜磷脂的结构, 达托霉素影响了多种重要细胞过程, 其中细胞壁合成受到的影响最大, 细胞壁的破损最终导致细胞裂解和细菌死亡。达托霉素长时间作用还影响细胞呼吸、细胞膜电势和细胞分裂。达托霉素对细胞生理的多种效应, 以及其作用靶点为液脂, 是其杀菌效率高和耐药性低的主要原因。虽然类似的液脂结构在葡萄球菌和链球菌等病原菌中也被观察到, 然而病原菌是否采用与枯草芽孢杆菌类似的机制尚不明确。
4 病原菌耐受达托霉素的机制
PG是达托霉素与细胞膜相互作用和发挥杀菌活性所必需的细胞成分。研究表明病原菌通过多种途径降低细胞膜中PG的含量, 如降低PG合成量, 增强PG转化为lysyl-PG或心磷脂的含量, 最终改变细胞膜的刚性和流动性来耐受达托霉素。与磷脂代谢相关的酶类参与了这一过程(图 2)[11, 12]。PgsA (PG合成酶)的功能缺失突变和MprF (lys-PG合成酶)的功能获得性突变导致PG含量降低。心磷脂合成酶(Cls)催化心磷脂的合成, 心磷脂需要消耗两分子PG, 导致PG含量的降低。Cls的功能获得性突变在金黄色葡萄球菌、屎肠球菌和粪肠球菌中被观察到。细胞壁结构的改变或细胞外膜适应机制也介导了达托霉素耐药。DltABCD突变增加了细胞壁磷壁酸丙氨酰化水平, 通过电荷排斥的方式限制达托霉素接触细胞膜。三组份信号转导系统LiaFSR和双组份信号转导系统WalKR的突变也与耐药性相关。缺失liaR可以恢复耐药菌株的敏感性, 虽然详细的分子机制尚不明确, 在粪肠球菌中可能由于调控了细胞膜心磷脂的分布[13]。WalKR参与细胞壁生物合成和稳态调节, 突变的WalKR调控细胞壁合成, 降低MurG定位错误对脂质Ⅱ合成的影响[14]。

Figure 2 Figure 2 Biosynthetic pathway of the major phospholipids in Gram-positive bacteria. PA: Phosphatidic acid; CDP-DAG: CDP-diacylglycerol; PS: Phosphatidylserine; PE: Phosphatidylethanolamine; G-3-P: Glycerol-3-phosphate; PG-P: Phosphatidylglycerol-3-P; PG: Phosphatidylglycerol; CL: Cardiolipin
5 构效关系
5.1 钙离子结合位点
在溶液中达托霉素与钙离子以1:1的比例结合后构象发生改变, 14~16个达托霉素分子聚集形成微团并接近细菌的细胞膜。当微团接近细胞膜的磷脂双分子层时解离形成单体, 达托霉素分子在脂肪酸尾链的引导下, 插入到磷脂双分子层, 在细胞膜中与钙离子以1:2的比例结合。达托霉素含有DXDG或EF-hand基序, 这种基序在钙调蛋白等钙离子结合蛋白中也被观察到[15, 16]。除了这几个氨基酸残基外, 其他氨基酸残基也参与与钙离子的相互作用。通过化学全合成达托霉素的衍生物[16, 17], 用枯草芽孢杆菌作为指示菌测定化合物的活性时发现, 在5 mmol·L-1钙离子浓度下, Tyr替换Kyn13或(2S, 3S)-mGlu替换(2R, 3S)-mGlu12导致活性显著降低。这种效应与钙离子浓度有关, 增加钙离子浓度能够补偿活性的降低, 暗示mGlu12和Kyn13可能参与了钙离子的结合, 而对作用机制无显著影响。
5.2 脂肪酸侧链的作用
研究表明, 脂肪酸侧链对于达托霉素活性是必需的。达托霉素衍生物的体内外抗菌活性随着碳链的增长而增加(直到C10), 碳链长度继续增长后, 部分衍生物体外活性有所增加, 然而体内抗菌活性并无改善[18]。碳链长度超过C11时衍生物毒性显著增加。在脂肪酸侧链和Trp1之间插入苯丙氨酸和类似的氨基酸对活性只有很小的影响。为了进行荧光研究将癸酰基替换为芘-丁酸等对活性的影响也很小[19]。这些结果表明, 脂肪酸侧链特定结构并不重要, 侧链的重要作用在于维持环脂肽的疏水性。
5.3 单个和组合氨基酸替换
达托霉素的全合成难度很大, 仅获得数个衍生物。利用组合生物合成对亚基、结构域或模块替换也仅获得30个衍生物。从这些单个和组合氨基酸替换的衍生物中, 获得了达托霉素构效关系的重要信息[20]。Asp7和Asp9对于达托霉素生物活性非常重要, L-Asn2/Glu12/Asn7和L-Asn2/Glu12/Asn9完全丧失了活性, 而L-Asn2/Glu12/Asn3和L-Asn2/Glu12的活性显著降低, 这表明Asp3和mGlu12对于活性非常重要, 但是非必需[15]。这个结果与基因敲除的结果一致, 敲除负责编码甲基转移酶的dptI, 导致mGlu12转变为Glu12, 衍生物的抗菌活性显著降低。综合比较用Trp、Ile及Asn代替Kyn13而得到的衍生物的抗菌活性时发现, Trp代替Kyn13不会增加抗菌活性, 而用Asn或Ile代替Kyn13导致生物活性降低[6]。D-Asn11被D-Ala或D-Ser代替, D-Ala8用D-Ser代替, 对活性没有显著的影响。而用D-Asn代替D-Ala8导致活性显著降低。Glu12/Trp13与达托霉素的活性相当, 而Ala7/Glu12/Trp13完全丧失了活性, 这表明Asp7对于达托霉素发挥作用是必需的, 这与组合生物合成得到的结果一致。
组合生物合成和化学全合成研究表明, Asp7和Asp9是发挥活性所必需的, Asp3、Thr4、mGlu12和Kyn13是发挥最佳活性所需的(表 1)。令人遗憾的是, 单氨基酸及多氨基替换得到的30个衍生物中未发现体外活性优于达托霉素的新化合物。
表1(Table 1)
Table 1 In vitro antibacterial of daptomycin analogues with amino acid substitutions[20]. aMinimum inhibitory concentration (MIC) of analog/MIC of daptomycin; bThis analogue contained (2S, 3S)-3-methylglutamate, ie, the diastereomer of the native amino acid residue; cDapa = L-diaminopropionic acid; d∞ indicates complete loss of activity Peptide Amino acid position MIC ratioa
Table 1 In vitro antibacterial of daptomycin analogues with amino acid substitutions[20]. aMinimum inhibitory concentration (MIC) of analog/MIC of daptomycin; bThis analogue contained (2S, 3S)-3-methylglutamate, ie, the diastereomer of the native amino acid residue; cDapa = L-diaminopropionic acid; d∞ indicates complete loss of activity Peptide Amino acid position MIC ratioa
Dap 1 2 3 4 5 6 7 8 9 10 11 12 13
Trp D-Asn Asp Thr Gly Orn Asp D-Ala Asp Gly D-Ser mGlu kyn 1
1 … L-Asn … … … … … … … … … … … 10
2 … L-Asn Asn … … … … … … … … Glu … 27
3 … L-Asn … … … … Asn … … … … Glu … ∞d
4 … L-Asn … … … … … … Asn … … Glu … ∞d
5 … L-Asn … … … … … … … … … Glu … 10
6 … L-Asn … … … … … … … … D-Asp Glu … > 100
7 … L-Asn … … … … … … … … … Glu Trp 33
8 … L-Asn … … … Lys … … … … … Glu Trp 33
9 … … … … … … … … … … … … Trp 2
10 … … … … … … … … … … … … Ile 4
11 … … … … … … … … … … … … Val 8
12 … … … … … … … … … … … Glu … 16
13 … … … … … … … … … … D-Ala … … 2
14 … … … … … … … … … … D-Asn … … 2
15 … … … … … … … D-Lys … … D-Asn … … 2
16 … … … … … … … … … … D-Asn Glu … 64
17 … … … … … … … … … … D-Asn … Ile 8
18 … … … … … … … D-Asn … … … … … 16
19 … … … … … … … D-Asn … … … Glu … 256
20 … … … … … … … D-Asn … … … … Ile 32
21 … … … … … … … D-Ser … … … … … 2
22 … … … … … … … … … … … mGlub … 40-50
23 … … … … … … … … … … … … Tyr 47
24 … … … … … … … … … … … Glu Trp 2-5
25 … … … Ser … … … … … … … Glu Trp > 100
26 … … … … … … Ala … … … … Glu Trp ∞d
27 … … … Dapac … … … … … … … Glu … 200
28 … … … Dapac … … … … … … … Glu Trp 100
29 Phe … … … … … … … … … … … … 4
30 Tyr … … … … … … … … … … … … > 32
Table 1 In vitro antibacterial of daptomycin analogues with amino acid substitutions[20]. aMinimum inhibitory concentration (MIC) of analog/MIC of daptomycin; bThis analogue contained (2S, 3S)-3-methylglutamate, ie, the diastereomer of the native amino acid residue; cDapa = L-diaminopropionic acid; d∞ indicates complete loss of activity
6 新型衍生物surotomycin
氨基酸替换未获得活性优于达托霉素的衍生物, 而脂肪酸侧链优化得到了对艰难梭菌(C. difficile)活性更佳的surotomycin (图 3)。艰难梭菌是一种非常重要的医院感染病原菌, 其感染占抗菌素相关腹泻的10%~25%, 占抗菌素相关肠炎的50%~75%[21]。现有的药物疗效差, 复发率高, 迫切需要开发新的治疗药物。虽然达托霉素在体外具有抗艰难梭菌活性, 但是其口服利用度很低, 胃肠道渗透性低, 难以用于CDI的治疗。脂肪酸侧链对于抗生素与细胞膜的结合有重要的作用, 影响抗生素活性和药理特性。构效关系研究表明, 脂肪酸侧链的亲脂性、链长度和关键芳香官能团的位置是活性优化的关键[22, 23]。通过大量合成衍生物和构效关系研究最终获得了surotomycin, 该化合物包含一个带12个碳原子侧链的(E)-3-(4-戊基苯基)丁-2-烯酰基。与达托霉素相比, surotomycin口服利用度高, 对艰难梭菌的MIC降低75%, 对达托霉素耐药肠球菌也有良好的杀菌效果。Surotomycin与达托霉素有类似的作用机制, 导致病原菌细胞膜去极化, 并不增加细菌细胞膜的通透性。耐药机制研究发现, 艰难梭菌的耐药突变发生在心磷脂合成酶Cls (D79N), 屎肠球菌的耐药突变包含Cls (R211L), 粪肠球菌的突变位于LiaS (M110I)和MprF2 (A29T), 这些都与达托霉素耐药位点类似, 耐药机制类似暗示作用机制相似[24]。

Figure 3 Figure 3 Acyl tail optimization to generate daptomycin derivative surotomycin
Surotomycin对艰难梭菌具有快速杀菌作用, 口服给药, 目前处于治疗CDI的Ⅲ期临床试验阶段[25-27]。相比于万古霉素, surotomycin具有更窄的抗菌谱, 对胃肠道正常革兰阳性菌微生物组的影响更小, 有助于降低复发率。临床前实验表明, surotomycin在仓鼠CDI模型中具有良好活性。临床试验表明, surotomycin (250 mg, 1天2次)导致更低的复发率。与体外活性结果一致, surotomycin在Ⅱ期临床试验中预防CDI复发的效果优于万古霉素, 耐受性好, 未发现安全问题。
7 其他进展
达托霉素仅对革兰阳性菌有活性, 而对革兰阴性菌没有活性。阴性菌细胞外膜是阻碍达托霉素与细胞膜相互作用的重要因素[28]。铁离子对于病原菌的生存很重要。病原菌为了从宿主中获得低浓度的铁离子, 通过分泌高亲和力的铁载体, 并以主动运输的方式转运铁离子进细胞[29]。将达托霉素与铁载体共价偶联, 通过铁载体递送达托霉素, 有效规避了细胞外膜的障碍, 从而发挥其强力杀菌作用。利用此思路构建的鲍曼不动杆菌铁载体-达托霉素偶联物获得强力体内和体外杀多重耐药菌活性(图 4), 耐药菌MIC从超过100 mol·L-1降低为0.4 mol·L-1 [30]。偶联物对铜绿假单胞菌、伯克氏菌或大肠杆菌没有活性, 这表明铁载体-达托霉素偶联物具有选择性。由此可知, 若将达托霉素与不同革兰阴性菌特异性铁载体偶联, 则有可能获得高度选择性的新型抗菌药物, 用于对抗日益严重的阴性菌耐药问题。此外, 通过构建长循环达托霉素脂质体能够实现高载药和对耐药金黄色葡萄球菌更持久的抑制效果, 增加达托霉素的肺部分布和提高小鼠感染模型中小鼠的存活率[31]。

Figure 4 Figure 4 Synthesis of synthetic siderophore-daptomycin conjugate with potent bactericidal activity against multidrug resistant Acinetobacter baumannii
8 展望
达托霉素的生物合成途径和作用机制已经基本明确, 而构效关系的阐明需要合成大量和多样的氨基酸替换衍生物来揭示每个氨基酸残基的功能。除了氨基酸残基外, 脂肪酸侧链也是影响抗生素抗菌谱和水溶性的重要因素, surotomycin的成功研发印证了这一点。构效关系的阐明将为理性设计新的衍生物用以解决达托霉素耐药性以及被肺部表面活性剂抑制活性等问题奠定坚实的基础。此外, 达托霉素与鲍曼不动杆菌特异性铁载体的偶联扩展达托霉素的抗菌谱, 使其有望成为控制革兰阴性菌所致感染的新选择, 更大地发挥达托霉素在感染性疾病控制中的价值。
参考文献
[1] Heidary M, Khosravi AD, Khoshnood S, et al. Daptomycin[J]. J Antimicrob Chemother, 2018, 73: 1–11. DOI:10.1093/jac/dkx349
[2] Pader V, Edwards AM. Daptomycin:new insights into an antibiotic of last resort[J]. Future Microbiol, 2017, 12: 461–464. DOI:10.2217/fmb-2017-0034
[3] Humphries RM, Pollett S, Sakoulas G. A current perspective on daptomycin for the clinical microbiologist[J]. Clin Microbiol Rev, 2013, 26: 759–780. DOI:10.1128/CMR.00030-13
[4] Cirioni O, Mocchegiani F, Ghiselli R, et al. Daptomycin and rifampin alone and in combination prevent vascular graft biofilm formation and emergence of antibiotic resistance in a subcutaneous rat pouch model of staphylococcal infection[J]. Eur J Vasc Endovasc Surg, 2010, 40: 817–822. DOI:10.1016/j.ejvs.2010.08.009
[5] Jiang H, Xiong M, Bi Q, et al. Self-enhanced targeted delivery of a cell wall- and membrane-active antibiotics, daptomycin, against staphylococcal pneumonia[J]. Acta Pharm Sin B, 2016, 6: 319–328. DOI:10.1016/j.apsb.2016.05.010
[6] Xie M, Wang F, Xu J, et al. Advances in daptomycin biosynthesis[J]. Microbiology China (微生物学通报), 2013, 40: 1754–1764.
[7] Beiras-Fernandez A, Vogt F, Sodian R, et al. Daptomycin:a novel lipopeptide antibiotic against Gram-positive pathogens[J]. Infect Drug Resist, 2010, 3: 95–101.
[8] Zhang Q, Chen Q, Zhuang S, et al. A MarR family transcriptional regulator, DptR3, activates daptomycin biosynthesis and morphological differentiation in Streptomyces roseosporus[J]. Appl Environ Microbiol, 2015, 81: 3753–3765. DOI:10.1128/AEM.00057-15
[9] Wang F, Ren NN, Luo S, et al. DptR2, a DeoR-type auto-regulator, is required for daptomycin production in Streptomyces roseosporus[J]. Gene, 2014, 544: 208–215. DOI:10.1016/j.gene.2014.04.044
[10] Muller A, Wenzel M, Strahl H, et al. Daptomycin inhibits cell envelope synthesis by interfering with fluid membrane microdomains[J]. Proc Natl Acad Sci U S A, 2016, 113: E7077–E7086. DOI:10.1073/pnas.1611173113
[11] Tran TT, Munita JM, Arias CA. Mechanisms of drug resistance:daptomycin resistance[J]. Ann N Y Acad Sci, 2015, 1354: 32–53. DOI:10.1111/nyas.2015.1354.issue-1
[12] Gomez Casanova N, Siller Ruiz M, Muñoz Bellido JL. Mechanisms of resistance to daptomycin in Staphylococcus aureus[J]. Rev Esp Quimioter, 2017, 30: 391–396.
[13] Reyes J, Panesso D, Tran TT, et al. A LiaR deletion restores susceptibility to daptomycin and antimicrobial peptides in multidrug-resistant Enterococcus faecalis[J]. J Infect Dis, 2015, 211: 1317–1325. DOI:10.1093/infdis/jiu602
[14] Howden BP, McEvoy CR, Allen DL, et al. Evolution of multidrug resistance during Staphylococcus aureus infection involves mutation of the essential two component regulator WalKR[J]. PLoS Pathog, 2011, 7: e1002359. DOI:10.1371/journal.ppat.1002359
[15] Grunewald J, Sieber SA, Mahlert C, et al. Synthesis and derivatization of daptomycin:a chemoenzymatic route to acidic lipopeptide antibiotics[J]. J Am Chem Soc, 2004, 126: 17025–17031. DOI:10.1021/ja045455t
[16] Lohani CR, Taylor R, Palmer M, et al. Solid-phase synthesis and in vitro biological activity of a Thr4——>Ser4 analog of daptomycin[J]. Bioorg Med Chem Lett, 2015, 25: 5490–5494. DOI:10.1016/j.bmcl.2015.10.072
[17] Lohani CR, Taylor R, Palmer M, et al. Solid-phase total synthesis of daptomycin and analogs[J]. Org Lett, 2015, 17: 748–751. DOI:10.1021/acs.orglett.5b00043
[18] Debono M, Abbott BJ, Molloy RM, et al. Enzymatic and chemical modifications of lipopeptide antibiotic A21978C:the synthesis and evaluation of daptomycin (LY146032)[J]. J Antibiot (Tokyo), 1988, 41: 1093–1105. DOI:10.7164/antibiotics.41.1093
[19] Muraih JK, Harris J, Taylor SD, et al. Characterization of daptomycin oligomerization with perylene excimer fluores-cence:stoichiometric binding of phosphatidylglycerol triggers oligomer formation[J]. Biochim Biophys Acta, 2012, 1818: 673–678. DOI:10.1016/j.bbamem.2011.10.027
[20] Taylor SD, Palmer M. The action mechanism of dap-tomycin[J]. Bioorg Med Chem, 2016, 24: 6253–6268. DOI:10.1016/j.bmc.2016.05.052
[21] Yin E, Wang X. Progress in the pathogenic mechanisms and treatments of human Clostridium Difficile infection[J]. Prog Modern Biomed (现代生物医学进展), 2013, 13: 5154–5158, 5040.
[22] Knight-Connoni V, Mascio C, Chesnel L, et al. Discovery and development of surotomycin for the treatment of Clostridium difficile[J]. J Ind Microbiol Biotechnol, 2016, 43: 195–204. DOI:10.1007/s10295-015-1714-6
[23] Yin N, Li J, He Y, et al. Structure-activity relationship studies of a series of semisynthetic lipopeptides leading to the discovery of surotomycin, a novel cyclic lipopeptide being developed for the treatment of Clostridium difficile-associated diarrhea[J]. J Med Chem, 2015, 58: 5137–5142. DOI:10.1021/acs.jmedchem.5b00366
[24] Adams HM, Li X, Mascio C, et al. Mutations associated with reduced surotomycin susceptibility in Clostridium difficile and Enterococcus species[J]. Antimicrob Agents Chemother, 2015, 59: 4139–4147. DOI:10.1128/AAC.00526-15
[25] Daley P, Louie T, Lutz JE, et al. Surotomycin versus vancomycin in adults with Clostridium difficile infection:primary clinical outcomes from the second pivotal, randomized, double-blind, phase 3 trial[J]. J Antimicrob Chemother, 2017, 72: 3462–3470. DOI:10.1093/jac/dkx299
[26] Chandorkar G, Zhan Q, Donovan J, et al. Pharma-cokinetics of surotomycin from phase 1 single and multiple ascending dose studies in healthy volunteers[J]. BMC Pharmacol Toxicol, 2017, 18: 24. DOI:10.1186/s40360-017-0123-z
[27] Cannon K, Byrne B, Happe J, et al. Enteric microbiome profiles during a randomized phase 2 clinical trial of surotomycin versus vancomycin for the treatment of Clostridium difficile infection[J]. J Antimicrob Chemother, 2017, 72: 3453–3461. DOI:10.1093/jac/dkx318
[28] Randall CP, Mariner KR, Chopra I, et al. The target of daptomycin is absent from Escherichia coli and other gram-negative pathogens[J]. Antimicrob Agents Chemother, 2013, 57: 637–639. DOI:10.1128/AAC.02005-12
[29] Huang W, Wilks A. Extracellular heme uptake and the challenge of bacterial cell membranes[J]. Annu Rev Biochem, 2017, 86: 799–823. DOI:10.1146/annurev-biochem-060815-014214
[30] Ghosh M, Miller PA, Mollmann U, et al. Targeted antibiotic delivery:selective siderophore conjugation with daptomycin confers potent activity against multidrug resistant Acinetobacter baumannii both in vitro and in vivo[J]. J Med Chem, 2017, 60: 4577–4583. DOI:10.1021/acs.jmedchem.7b00102
[31] Huang X, Wu J, Su T, et al. Long-circulating liposomal daptomycin enhances protection against systemic methicil-lin-resistant Staphylococcus aureus infection with improved therapeutic potential[J]. Acta Pharm Sin (药学学报), 2014, 49: 701–710.