2 University of Chinese Academy of Sciences, Beijing 100049, China
Yong Tao. Tel/Fax: +86-10-64807419; E-mail: taoyong@im.ac.cn
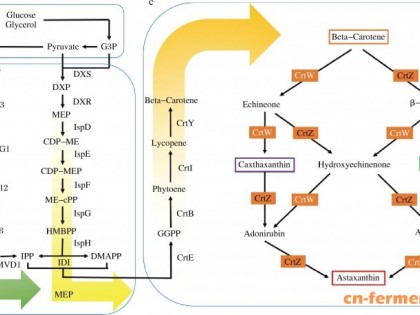
β-胡萝卜素是自然界中一种重要的天然色素,属于萜类脂溶性化合物。β-胡萝卜素具有很强的抗氧化活性,能够发挥多种重要的生理功能,其中包括体内维生素A的前体物质、预防肿瘤、延缓衰老、促进免疫功能[1-4]。在食品、医药、保健品、化妆品、饲料等多个领域,β-胡萝卜素均具有重要的应用价值和广泛的市场前景[5-6]。生产β-胡萝卜素的手段主要包括天然提取法、化学合成法、生物合成法。其中天然提取法通过从胡萝卜等植物中提取获得β-胡萝卜素,由于产率低、受季节影响等原因,无法满足市场需求。化学合成法虽然产量大、成本低,但具有毒化学物质残留等缺陷。相比而言,以微生物发酵为基础的生物合成法,易大规模生产,具有天然、安全等特点,是最具有发展前景的生产手段。
目前在微生物发酵法生产β-胡萝卜素的相关研究中,通过代谢工程手段改造β-胡萝卜素合成途径,从而提高β-胡萝卜素的生产水平一直是重要研究热点之一。其中大肠杆菌Escherichia coli因其易于培养和大规模发酵,同时具有完善的遗传改造手段,成为代谢工程合成β-胡萝卜素的最主要宿主菌株。与所有萜类化合物一样,β-胡萝卜素的生物合成途径依赖两种五碳前体——异戊烯焦磷酸(Isopentenyl pyrophosphate,IPP)和二甲基烯丙基焦磷酸(Dimethyl allyl pyrophosphate,DMAPP)。IPP与DMAPP通过类胡萝卜素(Carotenoids)合成基因簇编码蛋白的催化作用,经多步缩合反应生成β-胡萝卜素。在这一过程中,GGPP合成酶CrtE (Geranylgeranyl pyrophosphate synthase)、八氢番茄红素合成酶CrtB (Phytoene synthase)、八氢番茄红素脱氢酶CrtI (Phytoene desaturase)能够首先催化合成β-胡萝卜素的前体番茄红素。番茄红素进一步在番茄红素环化酶CrtY的作用下产生β-胡萝卜素。
目前,国内外β-胡萝卜素合成途径的改造在提高IPP及DMAPP前体供应方面开展了大量工作[7-10]。自然界中IPP和DMAPP主要有两条生物合成途径:以乙酰CoA为前体的甲羟戊酸途径(MVA途径)和以丙酮酸及3磷酸甘油醛为前体的2-C-甲基-D-赤藓糖醇-4-磷酸途径(MEP途径)。在大肠杆菌中构建异源MVA途径的工作以Keasling JD为代表[11-13]。Keasling JD等在大肠杆菌中引入了酿酒酵母的MVA途径基因,并将MVA途径分解成上游模块及下游模块分别进行优化。经过HMGR等关键靶点的基因替换与改造等手段,解除了HMG-CoA等中间体积累,实现了在大肠杆菌中利用MVA途径高水平合成紫穗槐等萜类产物。大肠杆菌中MEP途径的改造则以Stephanopoulos G为代表[14-15]。Stephanopoulos G等通过代谢流分析、随机改造等办法,验证了敲除gdhA、aceE、fdhF等对产物产量的提高。此外不同研究组通过改造pgi、gnd等靶点重构中央代谢途径,或sucAB、sdhAB和talB的过表达,均能改善MEP途径前体供应,从而有效提高β-胡萝卜素产量[16-17]。
相对前体供应途径改造而言,国内外对类胡萝卜素合成基因簇的优化工作还非常薄弱。大多数代谢工程合成β-胡萝卜素的工作中,直接采用了泛菌属Pantoea等少数微生物来源的基因簇合成β-胡萝卜素。本实验室前期研究中,通过挖掘不同来源的crtE、crtB和crtI基因,并利用不同强度的RBS对基因的表达水平同时进行调控,从而显著提高了番茄红素的合成水平[18]。以上结果表明,蛋白表达的精细调控对类胡萝卜素合成基因簇功能优化具有重要作用。在代谢工程传统蛋白表达调控改造中,通常采用随机突变或理性设计的办法构建不同强度的调控元件。但随机突变的调控元件强度无法控制;而理性设计的调控元件无法实现大规模的设计与构建,缺乏同时、高通量对多种因素进行优化的手段,极大影响了途径和模块优化的效率。本研究以先期优化的番茄红素合成模块(crtEBI)为基础,通过引入番茄红素环化酶基因(crtY)构建β-胡萝卜素合成模块(图 1),利用高通量的寡聚接头介导的DNA组装方法(Oligo-linker mediated assembly method,OLMA),从大规模RBS序列设计、改变基因来源等多个方面对crtY的表达进行微调控,实现对β-胡萝卜素合成模块的高通量优化,并阐明crtY表达水平对模块和代谢途径中其他基因功能以及目标产物的影响。寡聚接头介导的DNA组装方法是一种将小片段调控序列引入在寡聚接头的DNA组装文库构建方法,可同时调节大于50 bp和小于50 bp的影响因素[18]。该方法的独特性在于寡聚接头(小于50 bp因素)既可以被用作介导组装的“桥梁”,也可以被设计启动子、RBS等调控元件。由于寡聚接头易于合成,从而可以实现大规模的元件设计和组装。并且该方法可以同时对基因来源、位置等因素(大于50 bp因素)进行调控。通过将小于50 bp的影响因素被设计在化学合成的寡聚接头中,而大于50 bp的影响因素则通过标准载体引入,通过IIS型限制性内切酶获得相应的互补片段进行一步组装,大大提高了文库的构建效率和丰度。本研究将有助于阐明β-胡萝卜素合成模块表达的规律,为进一步提高β-胡萝卜素产量及其在工业中的应用打下基础。
 |
| 图 1 β-胡萝卜素合成模块的构建 Figure 1 Construction of β-carotene synthesis module. |
|
图选项
|
表达宿主菌Escherichia coli BW25113由本实验室保存;克隆宿主菌Escherichia coli Trans1-T1购自北京全式金生物技术有限公司;其他所有菌株和质粒见表 1。
| Strain/plasmid | Characteristics | Reference |
| Strain | ||
| Escherichia coli Trans1-T1 | F-φ80(lacZ) ΔM15ΔlacX74hsdR(rk-, mk+) ΔrecA1398 endA1 tonA | TransGen Biotech |
| Escherichia coli BW25113 | lacIqrrnBT14ΔlacZWJ16hsdR514 ΔaraBADAH33ΔrhaBADLD78 | Our lab |
| Pantoea agglomerans GMCC1.2244 | Pag crtY | Our lab |
| Erythrobacter sp. Nan35 | Ery crtY | Our lab |
| Escherichia coli DB 3.1 | F-gyrA462endA1Δ(sr1-recA) mcrB mrr hsdS20(rB-, mB-) supE44 ara14 galK2 lacY1 proA2 rpsL20(SmR) xyl-5λ-leu mtl1 | Life technology |
| CP00 | BW25113 (pYESKs, pSKPMIc) | This study |
| CP07 | BW25113 (pYESKs, pSKPMIc, pCP07) | This study |
| CP08 | BW25113 (pYESKs, pSKPMIc, pCP08) | This study |
| CP09 | BW25113 (pYESKs, pSKPMIc, p1-G3) | This study |
| CP10 | BW25113 (pYESKs, pSKPMIc, p2-A2) | This study |
| CP11 | BW25113 (pYESKs, pSKPMIc, p2-D5) | This study |
| CP12 | BW25113 (pYESKs, pSKPMIc, p2-G5) | This study |
| CP13 | BW25113 (pYESKs, pSKPMIc, p3-A5) | This study |
| plasmid | ||
| pLY116 | RSF1030 origin, kanR, lycopene module containing crtEBI operon | [18] |
| pYESKs | p15A origin, strR, MVA upper module | [19] |
| pSKPMIc | pSC101 origin, cmR, MVA lower module | [19] |
| pSC7s | pSC101 origon, StrR, tac promoter, tL3 terminator | Our lab |
| pFUS | ColE origin, SpeR, ccdB operon | Our lab |
| pHD | ColE origin, tetR | Our lab |
| pOSIP | ccdB | Our lab |
| pCP07 | pLY116, Pag crtY | This study |
| pCP08 | pLY116, Ery crtY | This study |
| pHD07 | pHD, Pag crtY | This study |
| pHD08 | pHD, Ery crtY | This study |
| pLY116-TC | pLY116, tac promoter, tL3 terminator, ccdB operon | This study |
紫外可见光分光光度计,UV2500,日本岛津公司(Shimadzu);凝胶成像系统,Bio-pro 200E,美国西蒙公司(SIM);电转化仪,Micro-pulser,美国伯乐公司(Bio-Rad);PCR仪,Mastercycler ProS,德国安本德公司(Eppendorf);微孔板恒温振荡器,MB100-4A,杭州州奥盛仪器有限公司;酶标仪,Synergy MX/SMATC,美国伯腾仪器公司(BioTek);5 L发酵罐,上海保兴生物设备工程有限公司;高效液相色谱(HPLC),Agilent 1220,美国安捷伦公司(Agilent)。
胰蛋白胨、酵母粉购自Oxoid公司;β-胡萝卜素、番茄红素标准品购自Sigma-Aldrich公司;DNA分子量标准购自Thermo Scientific公司;FastPfu DNA聚合酶购自北京全式金生物技术有限公司;Gibson克隆试剂盒、各种限制性内切酶、T4 DNA连接酶、磷酸化酶购自NEB (北京)公司;细菌基因组提取试剂盒、琼脂糖凝胶回收试剂盒购自Omega (北京)有限公司;其他常规生化试剂均为国产分析纯。
1.1.3 培养基与溶液LB (Luria-Bertani)培养基:1%胰蛋白胨,0.5%酵母粉,1% NaCl,其中LB固体培养基添加1.5%琼脂粉。
ZYM自诱导培养基:100 mL培养基中含酵母粉0.5 g,蛋白胨1 g,甘油0.5 g,葡萄糖0.05 g,阿拉伯糖0.2 g,Na2SO4 0.07 g,NH4Cl 0.27 g,MgSO4 0.024 g,KH2PO4 0.34 g,Na2HPO40.9 g,0.1 mL微量元素,pH 7.0[20]。
M9培养基:100 mL培养基中含终浓度2 mmol/L MgSO4,0.1 mmol/L CaCl2,20 mL 5×M9盐溶液(200 mL溶液中含Na2PO4·7H2O 12.8 g,KH2PO4 3.0 g,NaCl 0.5 g,NH4Cl 1.0 g),pH 7.0。
抗生素:培养基中根据菌株添加抗生素,不同抗生素的终浓度为:氯霉素17 μg/mL,卡那霉素50 μg/mL,链霉素50 μg/mL,四环素50 μg/mL。
1.2 方法 1.2.1 分子生物学构建各种分子克隆反应按照所用酶、试剂盒说明书进行,所用引物见表 2。其中crtY基因的PCR扩增以相应菌株的基因组为模板,细菌基因组采用细菌基因组提取试剂盒提取。PCR条件:94 ℃ 3 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 2 min,30个循环;72 ℃ 5 min。启动子、终止子片段分别以相应质粒为模板进行PCR扩增,PCR条件:94 ℃ 3 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 5 min。带有启动子、终止子的CrtY表达质粒或受体质粒的构建采用多片段Gibson组装方法,按照文献[21]方法进行。
| Primers | Sequence (5′-3′) |
| PagY-F | GTTTAACTTTCGCGCGCGTAACAGGAGGAATTAACCATGCCGCGGTATGATCTG |
| PagY-R | GATGCGTCATCGCCATTATCATTGCATCGCCTGTTG |
| EryY-F | GTTTAACTTTCGCGCGCGTAACAGGAGGAATTAACCATGAGCGAACGCGATTGC |
| EryY-R | GATGCGTCATCGCCATTATCATCGATGGTCCTTGCG |
| tac-F | CAGACGAAGAATCGTCGACACAGCTAACACCACGTC |
| tac-R | CGCGCGCGAAAGTTAAACAAAA |
| tL3-F | AATGGCGATGACGCATCCTCAC |
| tL3-R | GCGGAAGAGCGCTGCATGTTTCTACTGGTATTGGCA |
| HPagY-F | CATCATGCCTCCTCTAGAGGTCTCATTAAATGCCGCGGTATGATCTG |
| HPagY-R | GATTTTGGTCATGGGTGGCTCGAGGGTCTCAATACTCATTGCATCGCCTGTTG |
| HEryY-F | CATCATGCCTCCTCTAGAGGTCTCATTAAATGAGCGAACGCGATTGC |
| HEryY-R | GATTTTGGTCATGGGTGGCTCGAGGGTCTCAATACTCATCGATGGTCCTTGCG |
| ccd-F | GTTTAACTTTCGCGCGCGTAACAGGAGGAATTAACCCAGGTGAGACCAGGAGGAATTAACCATGCAGTTTAAGGTTTAC |
| ccd-R | GATGCGTCATCGCCATTAATACTGAGACCTTATATTCCCCAGAAC |
OLMA文库构建按照文献[18]进行。其中寡聚接头是通过将互补的两条单链oligo退火形成双链oligo。反应条件为终浓度各为10 μmol/L的单链oligo在90 ℃温育5 min,然后以每秒0.1 ℃的速度降至4 ℃。退火后将双链DNA稀释至终浓度100 nmol/L,将不同强度的寡聚接头等比例混合,利用磷酸化酶进行磷酸化反应,获得最终的寡聚接头片段。
1.2.3 重组菌株的诱导表达及生物转化挑取单克隆在新鲜的相应抗性的LB培养基中培养,待OD600值达到0.6-0.8,以1%的接种量转接到含有相应抗性的ZYM自诱导培养基中培养12-14 h,诱导MVA途径基因的表达。4 ℃、4 000 r/min条件下离心,弃去培养基,然后用M9培养基和1%葡萄糖的转化液重悬菌体至10 OD/L,37 ℃、220 r/min摇床进行生物转化20 h。测定转化后β-胡萝卜素产量水平。
1.2.4 β-胡萝卜素、番茄红素的测定对生物转化后的菌体细胞测定菌体OD600,取1×108 CFU细胞的培养液离心收集菌体,双蒸水洗涤菌体1次,于菌体中加入1 mL丙酮,冰上萃取2 h后,13 000 r/min离心10 min,吸取上清液。β-胡萝卜素的测定用分光光度计扫描340-550 nm的吸收光谱,并读取454 nm处的特征吸收峰值,再根据标准曲线计算β-胡萝卜素的产量,实验设计3次重复。用β-胡萝卜素标准品制定标准曲线。β-胡萝卜素、番茄红素产物的定性分析通过HLPC方法测定上述样品,使用Phenomenex Luna 5u C8(2) 100A色谱柱,流动相为水+丙酮(体积比为20:80),以β-胡萝卜素、番茄红素标准品标定。
1.2.5 OLMA文库的筛选平板筛选,37 ℃培养12 h后将黄色克隆挑取于含有200 μL LB培养基的96孔深孔板中,于37 ℃、800 r/min的微孔板恒温振荡器中培养10 h后按1:40的比例接种于200 μL含诱导物的ZYM自诱导培养基的96孔深孔板中,于37 ℃、800 r/min的振荡器中恒温培养16 h。将诱导后的培养液稀释20倍,用酶标仪测定OD600。同时取20 μL的培养液,4 000 r/min离心10 min收集菌体,加入400 μL乙醇-丙酮混合液(体积比4:1)冰上萃取2 h。萃取完成后12 000 r/min离心10 min,吸取200 μL上清液于96孔微孔板中(平底),用酶标仪读取454 nm处的吸光值,计算吸光值与菌体OD600的比值,利用该值评判相应克隆的β-胡萝卜素合成能力。
1.2.6 中间产物分析对生物转化后的菌体细胞,测定菌体OD600,取5×109CFU细胞的培养液离心收集菌体,用-80 ℃预冷的甲醇重悬,超声后离心收取上清,经冷冻干燥制备样品。样品委托清华大学生物医学测试中心UPLC-MS/MS分析检测。
1.2.7 5 L发酵水平生产β-胡萝卜素挑取单克隆在相应抗性的LB培养基中培养过夜,5%的接种量接入到装液量为100 mL的LB培养基中,培养5 h,再接入装液量为2 L的5 L发酵罐中,发酵培养基为(/L):20 g葡萄糖,5 g酵母粉,0.6 g MgSO4,9 g KH2PO4,4 g (NH4)2HPO4,及微量元素。生长温度控制在37 ℃,pH控制在7.0,空气流速为4 L/min,发酵过程中DO控制在30%,发酵过程中恒速补料。
2 结果与分析 2.1 产β-胡萝卜素大肠杆菌菌株的构建根据NCBI序列设计引物PagY-F/PagY-R和EryY-F/EryY-R,分别以提取的成团泛菌Pantoea agglomerans CGMCC1.2244和赤杆菌Erythrobacter sp. Nan35的基因组总DNA为模板进行PCR扩增,获得Pag-crtY和Ery-crtY基因片段。分别用tac-F/tac-R和tL3-F/tL3-R引物,以质粒pSC7s为模板,分别扩增出tac启动子片段tac和终止子片段tL3。采用SalⅠ单酶切本实验室之前构建的含有番茄红素合成基因簇crtEBI的载体pLY116,回收载体大片段后,采用Gibson方法分别与tac/Pag-crtY/tL3或tac/Ery-crtY/tL3进行多片段连接,筛选阳性克隆,得到具有完整合成途径的β-胡萝卜素合成模块质粒pCP07和pCP08。将pCP07及pCP08质粒和分别含有MVA上游途径基因的pYESKs质粒和MVA下游途径基因的pSKPMIc质粒共转,获得具有合成β-胡萝卜素能力的工程菌株CP07及CP08 (图 2)。
 |
| 图 2 在番茄红素合成模块的基础上引入CrtY及OLMA文库 Figure 2 Manipulation of CrtY and OLMA library based on lycopne synthesis module. |
|
图选项
|
为了验证CP07和CP08的功能,将CP07和CP08接种于ZYM培养基中诱导表达β-胡萝卜素合成途径蛋白,收集菌体后重悬于20 mL含有1%葡萄糖的M9培养基中,摇瓶生物转化20 h。收集菌体后用丙酮萃取产物,检测β-胡萝卜素含量,以无β-胡萝卜素合成模块质粒的菌株CP00为对照。结果表明(图 3,7C),经转化后CP07和CP08的色素产物均为单一的β-胡萝卜素,β-胡萝卜素合成水平分别达到约8.79 mg/g DCW及8.08 mg/g DCW。
 |
| 图 3 CP07和CP08的β-胡萝卜素合成水平 Figure 3 β-carotene production of CP07 and CP08. |
|
图选项
|
为了实现β-胡萝卜素合同途径中基因表达的平衡,并进一步提高β-胡萝卜素的合成水平,采用寡聚接头介导DNA组装(OLMA)文库构建的策略对crtY的表达水平进行微调控,OLMA文库的设计和构建过程如图 2和图 4所示。
 |
| 图 4 利用OLMA优化β-胡萝卜素合成模块的设计与构建 Figure 4 Design and construction of OLMA library to optimize β-carotene synthesis module. |
|
图选项
|
选择Pag-crtY及Ery-crtY两种crtY基因以增加文库的多样性。分别采用引物HPagY-F/ HPagY-R和HEryY-F/HEryY-R,以质粒pCP07和pCP08为模板,扩增HPag-crtY和HEry-crtY基因片段。采用AvaⅠ和XbaⅠ双酶切标准载体pHD,以Gibson方法分别与HPag-crtY和HEry-crtY连接,筛选阳性克隆,获得用于OLMA文库构建的Pag-crtY及Ery-crtY基因供体质粒pHD07和pHD08。其中crtY基因序列两端分别带有BsaⅠ酶切位点,上下游酶切粘性末端分别为TTAA和GTAT。
进一步对于构建文库所用的RBS序列优化。根据文献中的设计原则,以Pag-crtY及Ery-crtY的序列为基础,利用RBS Calculator软件计算出不同强度的RBS[18],并选取了50个可覆盖较大强度范围(~100-50 000)的RBS,将RBS核心序列、及上下游的粘性末端CAGG与TTAA设计在寡聚接头序列中。并制备磷酸化的寡聚接头。
以pLY116为基础构建受体质粒。利用引物ccd-F/ccd-R,以质粒pOSIP为模板,扩增编码毒蛋白的ccdB基因片段,采用Gibson方法与tac/tL3及经SalⅠ单酶切的pLY116进行多片段连接,转化感受态细胞大肠杆菌DB3.1中,筛选阳性克隆,获得受体质粒pLY116-TC。该质粒由于含有ccdB毒蛋白无法在DB3.1以外的正常大肠杆菌细胞中生长,从而消除文库构建中的假阳性。其中ccdB基因序列两端分别带有BsaⅠ酶切位点,上下游酶切粘性末端分别为CAGG和GTAT。
将受体质粒pLY116-TC、两种供体质粒及双链寡聚接头在含有BsaⅠ和T4 DNA连接酶的反应体系中实现一步组装,电转化进入T1感受态细胞。提取混合质粒,并转入含有pYESKs质粒和pSKPMIc质粒的BW25113感受态细胞中,用于下一步筛选。
2.3 OLMA文库的筛选和鉴定以CP07及CP08为对照,随机挑取33个克隆于96孔深孔板中,经LB培养基培养、ZYM培养基自诱导后,乙醇-丙酮检萃取,并用酶标仪检测单位菌体的β-胡萝卜素含量。结果如图 5A所示,β-胡萝卜素产量为0.47-1.04。根据相对产量数值分布看,所构建的OLMA文库可以引起正态分布的β-胡萝卜素产量变化,说明该手段能够有效地调控crtY的活性从而得到不同的β-胡萝卜素产量(图 5B,C)。
 |
| 图 5 利用96孔板对OLMA文库的筛选和鉴定 Figure 5 Screen and identification of OLMA library using 96 plates. (A) 33 clones were picked randomly and relative β-carotene production was determined. (B) Comparison of the relative β-carotene production of 33 strains with CP07 and CP08. (C) Statistics of strains. |
|
图选项
|
挑选克隆中有5株β-胡萝卜素产量明显超过CP07及CP08。选择相应的克隆命名为CP09-CP13,并进行复筛。将上述菌株在含有ZYM培养基的摇瓶中诱导后进行生物转化,检测β-胡萝卜素产量。结果表明,5株菌的β-胡萝卜素产量分别为16.89、16.89、15.79、18.89和16.96 mg/g DCW,其中最高的CP12的β-胡萝卜素产量比对照菌株CP07和CP08分别提高了65%和72% (图 6B)。同时对ZYM培养基中诱导后菌体量进行比较,发现CP09-CP13在诱导后的菌体量均比CP07和CP08有明显提高(图 6A)。
 |
| 图 6 摇瓶中高产菌株的生物量(A)及β-胡萝卜素产量(B) Figure 6 Biomass (A) and β-carotene production (B) of the 5 high-yield strains. |
|
图选项
|
为了进一步确定微调控对crtY表达的作用,对上述菌株进一步对序列进行分析。5株菌的RBS序列及相对强度见表 3。
| Strains | RBS sequence | RBS intensity | crtY |
| CP09 | ATGAATGTATCAATTGAGGT TTAA | 1 147 | Pag crtY |
| CP10 | CTATGAATTGAGCTAAGAGGTGAAA | 2 059 | Pag crtY |
| CP11 | GGAAACCCTCAGGAGGTAAACCA | 46 327 | Pag crtY |
| CP12 | ACTAAAACTTGAATGAGGAAATT | 3 088 | Pag crtY |
| CP13 | ATGAATGTATCAATTGAGGTTTAA | 1 147 | Ery crtY |
为了进一步研究crtY表达调控对代谢途径的影响,选取CP12的生物转化后菌体进行中间产物分析,检测菌体细胞中MVA代谢中间产物HMG-CoA (3-hydroxy-3-methyl glutaryl coenzyme A)以及IPP/DMAPP的含量。结果表明(图 7),与优化前菌株CP07相比,CP12的HMG-CoA含量提高约30%,而同时IPP/DMAPP的含量下降了4倍。采用HPLC检测转化后CP12的色素产物类型,结果表明CP12的色素产物与CP07、CP08一致,均只含有β-胡萝卜素(图 7C)。
 |
| 图 7 菌株的代谢中间产物分析 Figure 7 Analysis of metabolic intermediates of strains. (A) LC-MS/MS analysis of relative HMG-CoA concentrations. (B) LC-MS/MS analysis of relative IPP/DMAPP concentrations. (C) HPLC analysis of β-carotene and lycopene. |
|
图选项
|
为了进一步验证所筛选的高产β-胡萝卜素菌株的生产能力,选取CP12菌株在5 L发酵罐水平上进行发酵及β-胡萝卜素的生产(图 8)。开始发酵后控制菌体生长至10 h时,加入20 mL 20%的阿拉伯糖诱导物。15 h时开始检测β-胡萝卜素产物积累。结果发现发酵30 h后菌体浓度可达150 OD/L,同时β-胡萝卜素产物积累明显上升。至发酵50 h后,菌体浓度可达200 OD/L。β-胡萝卜素产量约为1.9 g/L。
 |
| 图 8 CP12的β-胡萝卜素发酵时间曲线 Figure 8 Time curve of β-carotene fermentation of CP12. |
|
图选项
|
利用代谢工程手段在微生物中改造目标产物的合成途径,通常需要解决多种异源蛋白的同时表达问题,从而解除途径中多个关键性限制瓶颈,提高目标产物合成水平。之前的研究表明利用OLMA方法对番茄红素合成模块中的crtE、crtB、crtI协同表达进行优化能够显著提高番茄红素的合成水平,证明蛋白表达的精细控制对类胡萝卜素合成基因簇具有重要作用[18]。在β-胡萝卜素合成模块中,番茄红素环化酶是一种需要结合FAD作为配体的环化酶[22],具有典型的跨膜结构域[23]。这一类蛋白在大肠杆菌中的可溶性表达往往具有很大的挑战性,之前的报道也证实番茄红素环化酶容易在大肠杆菌中形成不溶性包涵体[22]。本研究尝试以OLMA方法为基础对番茄红素环化酶的活性进行高效率的优化。结果表明优化后的菌株CP09-CP13的β-胡萝卜素产量均能提高60%以上。通过对高产菌株中RBS强度分析,5株高产菌株中有4株采用了低强度的RBS (预测强度为1147-3088),这一结果表明降低表达强度有利于番茄红素环化酶的活性。由于crtY及β-胡萝卜素合成模块其他基因的表达产物在CP07-CP13的菌株中表达量少,很难用SDS-PAGE分析(结果未列出),因此进一步用代谢中间体的积累分析相关途径和模块的功能。相关结果证实在降低番茄红素环化酶表达强度的工程菌株中,能够明显减少IPP/DMAPP的中间体积累,说明β-胡萝卜素合成模块的活性得到了提高。菌株CP07和CP12中色素产物均仅以β-胡萝卜素的形式存在,表明crtY在两个菌株中的表达量均足够将番茄红素转化为β-胡萝卜素。因此可以推测在菌株CP12中crtY优化对β-胡萝卜素产量的提高可能是由于平衡了crtY与crtEBI之间的表达,并促进了crtEBI的功能。值得注意的是,菌株CP09-CP13不仅β-胡萝卜素产量明显提高,在合成途径诱导之后菌体生长也比CP07和CP08明显提高,生长提高的原因尚不清楚。文献报道证实[11-12, 24],MVA途径的代谢中间体包括HMG-CoA、甲羟戊酸、IPP/DMAPP等均对细胞的生长具有毒性。在本研究中,菌株CP12中的IPP/DMAPP水平比未经优化的对照菌株CP07减少了4倍,暗示高产菌株生长的提高可能是由于代谢途径畅通后,解除了IPP/DMAPP的生长抑制引起的。
在微生物代谢工程改造中,只有异源途径、模块中不同异源蛋白的表达处于相对平衡的状态时,才能使模块的功能以及模块间的“适配性”、模块与宿主细胞间的“适配性”达到最优状态。本研究以β-胡萝卜素模块为研究对象,通过对模块中关键性的番茄红素环化酶的优化实现了异源蛋白表达的平衡,为高产β-胡萝卜素菌株的进一步优化及工业化应用打下基础。
| [1] | Chew BP. Vitamin A and β-carotene on host defense. J Dairy Sci, 1987, 70(12): 2732–2743. DOI: 10.3168/jds.S0022-0302(87)80346-6 |
| [2] | Olson JA. Provitamin A function of carotenoids: the conversion of beta-carotene into vitamin A. J Nutr, 1989, 119(1): 105–108. |
| [3] | Hughes DA, Finglas PM, Wright AJA, et al. Dietary beta-carotene supplementation modulates the production of tumour necrosis factor-α by human monocytes. Biochem Soc Trans, 1996, 24(3): 387S. DOI: 10.1042/bst024387s |
| [4] | Cui BK, Liu S, Wang QB, et al. Effect of β-carotene on immunity function and tumour growth in hepatocellular carcinoma rats. Molecules, 2012, 17(7): 8595–8603. |
| [5] | Yamano S, Ishii T, Nakagawa M, et al. metabolic engineering for production of β-carotene and lycopene in Saccharomyces cerevisiae. Biosci Biotechnol Biochem, 1994, 58(6): 1112–1114. DOI: 10.1271/bbb.58.1112 |
| [6] | Sandmann G. Carotenoid biosynthesis and biotechnological application. Arch Biochem Biophys, 2001, 385(1): 4–12. DOI: 10.1006/abbi.2000.2170 |
| [7] | Yoon SH, Park HM, Kim JE, et al. Increased β-carotene production in recombinant Escherichia coli harboring an engineered isoprenoid precursor pathway with mevalonate addition. Biotechnol Progr, 2007, 23(3): 599–605. |
| [8] | Yoon SH, Lee SH, Das A, et al. Combinatorial expression of bacterial whole mevalonate pathway for the production of β-carotene in E. coli. J Biotechnol, 2009, 140(3/4): 218–226. |
| [9] | Yan GL, Wen KR, Duan CQ. Enhancement of β-carotene production by over-expression of HMG-CoA reductase coupled with addition of ergosterol biosynthesis inhibitors in recombinant Saccharomyces cerevisiae. Curr Microbiol, 2012, 64(2): 159–163. DOI: 10.1007/s00284-011-0044-9 |
| [10] | Ye LJ, Zhang CZ, Bi CH, et al. Combinatory optimization of chromosomal integrated mevalonate pathway for β-carotene production in Escherichia coli. Microb Cell Fact, 2016, 15(1): 202. DOI: 10.1186/s12934-016-0607-3 |
| [11] | Martin VJJ, Pitera DJ, Withers ST, et al. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nat Biotechnol, 2003, 21(7): 796–802. DOI: 10.1038/nbt833 |
| [12] | Pitera DJ, Paddon CJ, Newman JD, et al. Balancing a heterologous mevalonate pathway for improved isoprenoid production in Escherichia coli. metab Eng, 2007, 9(2): 193–207. DOI: 10.1016/j.ymben.2006.11.002 |
| [13] | Ma SM, Garcia DE, Redding-Johanson AM, et al. Optimization of a heterologous mevalonate pathway through the use of variant HMG-CoA reductases. metab Eng, 2011, 13(5): 588–597. DOI: 10.1016/j.ymben.2011.07.001 |
| [14] | Alper H, Jin YS, Moxley JF, et al. Identifying gene targets for the metabolic engineering of lycopene biosynthesis in Escherichia coli. metab Eng, 2005, 7(3): 155–164. DOI: 10.1016/j.ymben.2004.12.003 |
| [15] | Alper H, Fischer C, Nevoigt E, et al. Tuning genetic control through promoter engineering. Proc Natl Acad Sci USA, 2005, 102(36): 12678–12683. DOI: 10.1073/pnas.0504604102 |
| [16] | Li C, Ying LQ, Zhang SS, et al. Modification of targets related to the Entner-Doudoroff/pentose phosphate pathway route for methyl-D-erythritol 4-phosphate-dependent carotenoid biosynthesis in Escherichia coli. Microb Cell Fact, 2015, 14(1): 117. DOI: 10.1186/s12934-015-0301-x |
| [17] | Zhao J, Li QY, Sun T, et al. Engineering central metabolic modules of Escherichia coli for improving β-carotene production. metab Eng, 2013, 17: 42–50. DOI: 10.1016/j.ymben.2013.02.002 |
| [18] | Zhang SS, Zhao XL, Tao Y, et al. A novel approach for metabolic pathway optimization: oligo-linker mediated assembly (OLMA) method. J Biol Eng, 2015, 9(1): 23. DOI: 10.1186/s13036-015-0021-0 |
| [19] | Feng F, Xu Y, Tao Y, et al. Improving isoprene production by engineered heterologous mevalonate pathway in Escherichia coli. Chin J Biotech, 2015, 31(7): 1073–1081. (in Chinese). 冯凡, 许杨, 陶勇, 等. 提高大肠杆菌通过MVA途径合成异戊二烯. 生物工程学报, 2015, 31(7): 1073-1081. |
| [20] | Studier FW. Protein production by auto-induction in high-density shaking cultures. Protein Expres Purif, 2005, 41(1): 207–234. DOI: 10.1016/j.pep.2005.01.016 |
| [21] | Gibson DG, Young L, Chuang RY, et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods, 2009, 6(5): 343–345. DOI: 10.1038/nmeth.1318 |
| [22] | Yu QJ, Schaub P, Ghisla S, et al. The lycopene cyclase CrtY from Pantoea ananatis (formerly Erwinia uredovora) catalyzes an FADred-dependent non-redox reaction. J Biol Chem, 2010, 285(16): 12109–12120. DOI: 10.1074/jbc.M109.091843 |
| [23] | Peck RF, Johnson EA, Krebs MP. Identification of a lycopene β-cyclase required for bacteriorhodopsin biogenesis in the Archaeon Halobacterium salinarum. J Bacteriol, 2002, 184(11): 2889–2897. DOI: 10.1128/JB.184.11.2889-2897.2002 |
| [24] | Pfleger BF, Pitera DJ, Smolke CD, et al. Combinatorial engineering of intergenic regions in operons tunes expression of multiple genes. Nat Biotechnol, 2006, 24(8): 1027–1032. DOI: 10.1038/nbt1226 |