【摘要】 大环内酯类抗生素结构中的糖基基团在抑菌、生产菌自我保护和调控大环内酯类抗生素内酯环合成方面有着重要的生物学功能。了解糖基的合成途径、糖基转移酶的结构特点和糖基生物学功能对研制新结构抗生素,解决当前日益严重的致病菌耐药性问题起着重要的指导作用。通过基因工程的方法改造糖基,已经合成了一系列具有抗菌活性的新结构化合物。本文就目前所了解的糖基合成途径、糖基转移酶的结构特点和糖基重要生物学功能做一综述,并简单介绍基因工程改造糖基合成新结构抗生素的研究进展。
【关键词】 大环内酯类抗生素 糖基 生物学功能 结构改造
Glycosyl synthesis and biofunction of macrolide antibiotics
ABSTRACT The review summarized synthesis pathway of glycosyl linked to macrolide antibiotics, the structure feature of their glycosytransferase and glycosyl biofunction of macrolide antibiotics, and briefly introduced progress in modifying glycosyl to synthsize novel structure antibiotics by genetic engineering.
KEY WORDS Macrolide antibiotics; Glycosyl; Biofunction; Structure modification
大环内酯类抗生素是一类根据其结构特点命名的广谱抗生素,由多元内酯环和后修饰基团相连而成。根据内酯环碳原子数目不同,可分为12~16元环抗生素,其中,在临床上最重要、最常见大环内酯类抗生素有12元环的酒霉素(methymycin)、苦霉素(pikro?mycin),14元环的红霉素(erythromycin)、竹桃霉素(oleandomycin),15元环的阿奇霉素(azithromycin)和16元环的螺旋霉素(spiramycin)、泰洛星(tylosin)等。大环内酯类抗生素合成过程中要经过一些后修饰,如羟基化、甲基化和糖基化等[1~4],这些后修饰基团都有着重要的生物学功能,其中,通过糖基化后修饰连接于内酯环上的糖基特别是脱氧糖基参与靶位点的分子识别作用,是抗生素表现抗菌活性的必要组成成分。近几年,随着分子生物学技术的发展,在研究抗生素某些合成基因功能的时候,又发现了内酯环上连接的糖基的其它生物学功能,虽然目前还不清楚其中有些糖基生物学功能的确切机理, 但了解了这些生物学功能对解决当前日益严重的致病菌耐药性问题,研制新结构抗生素起着重要的指导作用。通过基因工程改造糖基合成一系列具有抗菌活性新结构物质的研究已取得了一定进展。本文就目前所了解的大环内酯类抗生素上连接糖基的合成途径,糖基转移酶的结构特点,糖基在抑菌、生产菌自我保护和调控大环内酯类抗生素内酯环合成方面的生物学功能做一综述,简单介绍基因工程改造糖基合成具有抗菌活性新结构物质的研究进展。
1 糖基合成途径及其转移酶的结构特点
连接于大环内酯类抗生素内酯环上的后修饰糖基基团大部分属于6?脱氧己糖(6?deoxyhexose,6?DOHS),以2,6?脱氧己糖(2,6?dideoxyhexose)居多,迄今至少有80种不同的6?DOHS被报道[5~7],其中大多数参与构成不同的抗生素。在临床上常见的大环内酯类抗生素上连接的脱氧糖基有D?红霉糖胺(desasomine)、L?夹竹桃糖L?oleandrose)和D?碳霉糖(D?mycaminose)等等(表1)。 目前这些脱氧糖生物合成过 表1 几种常见大环内酯类抗生素上连接的脱氧糖
糖基添加到内酯环上需要糖基转移酶的作用。许多大环内酯类抗生素的糖基转移酶具有底物灵活性的特点。根据其针对的底物灵活性不同,可把糖基转移酶分为对脱氧糖具有灵活性、对底物内酯环具有灵活性、对脱氧糖和内酯环都具有灵活性三种。已报道对底物糖基具有灵活性的糖基转移酶有OleG2、对底物内酯环具有灵活性的糖基转移酶有DesvII、TylM2、MycB等[10~12],而糖多孢红霉菌中的eryCIII编码的红霉糖胺糖基转移酶却具有相对严格的底物特异性[13]。研究发现这些糖基转移酶在一级结构序列上几乎不具相似的保守性,却具有高级结构上相似性:有双叶状的两个结合域,一个是具有亲核性内酯环骨架的结合位点,另一个是具有亲电性NDP?糖的结合位点[14]。转移糖基的过程也就是NDP?糖捕获内酯环的过程。在利用整合生物合成的方法合成新结构抗生素方面,具有底物灵活性糖基转移酶是一种重要的工具。近年来随着生物技术发展,对其编码基因的深入了解,可以通过突变、敲除等方法来改变糖基转移酶结合域的特异性,产生更多具有底物灵活性转移酶,为合成新结构抗生素提供有力工具[15]。
2 糖基的生物学功能
2.1 活化内酯环的作用
大环内酯类抗生素内酯环本身没有抑菌活性,要经过后修饰基团如甲基、羟基、糖基等活化后才具有抗菌活性[1~4]。其中糖基尤其氨基糖基是活化内酯环关键性基团,在抑菌方面起着关键性作用(以红霉素和泰洛星为例说明)。
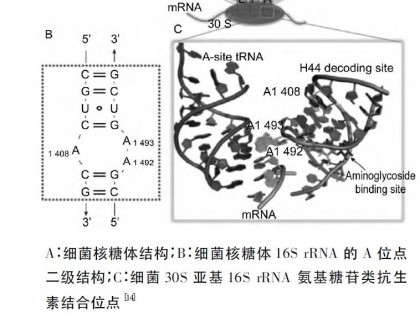
14元环的红霉素主要通过与核糖体50S亚型的23S rRNA的domainV结构域A2058位点结合,促进转录阶段的肽早熟性分离和阻碍新生肽的释放来抑制细菌生长。与A2058结合的是红霉素内酯环C?5位红霉糖胺的2?OH[16]。A2058的残基N1离domainV中二脱氧?D(L)?己糖合成路径细菌生长。与A2058结合的是红霉素内酯环C?5位红霉糖胺的2?OH[16]。A2058的残基N1离domainV中心A2451位点大约有15nm的距离,A2451是肽酰转移酶催化中心的结合位点,当合成的新生肽链长度达到3~5个氨基酸时,红霉素通过与A2058位置结合阻断肽链的延伸,促进转录阶段的肽早熟性分离。位点A2058又是靠近肽酰转移酶的底部,并且在新生肽释放隧道的入口处,结合在此位点上红霉素分子又会有效地阻碍新生肽的释放。
16元环泰洛星作用机理是通过影响肽酰转移酶的活性来阻碍新生肽的肽键形成,抑制细菌的生长。泰洛星内酯环上连接的二糖上的mycarose是其活性所必不可少的基团,缺乏mycarose会使泰洛星失去抑制细菌蛋白合成的能力[17]。研究发现mycarose的结合位点是U2506[18],U2506与肽酰转移酶(tRNAPhe)3′端的2N3A76在P位点上的结合位点横穿连接[19],而U2506又距离domainV中心位点A2541(A2451是肽酰转移酶催化中心的结合位点)只有大约90nm。由于空间距离上的靠近,使内酯环上连接的另外一个糖基mycinose直接干涉了肽酰转移酶的反应,阻碍新生肽肽键形成,抑制细菌生长。
2.2 调节内酯环合成的作用
泰乐酮(tylactone)内酯环上连接的碳霉糖糖基具有调节内酯环合成的作用,是1997年Fish等[20]在研究泰洛星产生菌弗氏链霉菌(S.fradiae)中的碳霉糖转移酶基因tylM2功能时候发现的。Fish发现敲除基因tylM2的弗氏链霉菌不能合成内酯环泰乐酮,当只有外加碳霉糖基化的泰乐酮或其它连有碳霉糖基的抗生素后,弗氏链霉菌才恢复合成泰乐酮的能力。在弗氏链霉菌的染色体上,tylM2处于泰乐酮合成基因tylG和调控基因tylR的下游,基因tylM2的敲除可能不会直接影响tylG和tylR的表达,但可能会影响调控基因tylR表达的调控因子TylR的功能。推测碳霉糖基化的泰乐酮可能是作为调控蛋白TylR的一个辅因子,与调控蛋白结合,共同调节tylG表达[20,21],但是对于具体调控机理至今未有文献报道。
同样的调控机理可能也存在大环内酯类抗生素spinosad的生产菌S.spinosa中,鼠李糖(rhamnose)是第一个连接到spinosad内酯环上的糖基,当敲除L?鼠李糖基(L?rhamnosyl)转移酶基因spnG后,生产菌没有spinosad和其它前体的合成[22],同样,当加入外源的糖基化前体后,菌株恢复合成spinosad和其内酯环的能力,具体的调控机理也不清楚。
2.3 失活抗生素的作用
某些大环内酯类抗生素生产菌为防止抗生素对自身致死性作用,进行自我保护,需要具有失活抗生素的作用。就目前所了解,这种失活抗生素作用的机制有三种:①对细菌核糖体上的抗生素结合位点进行甲基化或磷酸化等修饰机制;②加速抗生素外排机制;③糖基化失活抗生素机制。其中,第三种是生产菌对抗生素内酯环上6?脱氧己糖做进一步糖基化修饰以失活抗生素。这种自我保护机制最初在淡青紫链霉菌(S.lividans)中发现[23]。之后又在竹桃霉素的产生菌抗生素链霉菌(S.antibioticus)、酒霉素和新酒霉素(neome?thymycin)产生菌委瑞内拉链霉菌(S.venezuelae)中发现[24]。现已知道,在32种生产聚酮类抗生素的链霉菌中有15种存在这种抗性机制[25]。下面就以抗生素链霉菌为例来说明这种作用机制。
在抗生素链霉菌中,糖基化作用是以UDP?葡萄糖为糖基的供体,在位于细胞质中的糖基转移酶OleI作用下,对竹桃霉素内酯环C?5位单糖夹竹桃糖的2?OH进行糖基化修饰。2?OH是与核糖体靶位点结合的功能性基团,修饰后葡萄糖基阻碍了2?OH与靶位点的结合,致使竹桃霉素失去活性。同时,分泌到细胞外的竹桃霉素在胞外β?糖苷脱糖基酶OleR作用下脱去2?OH上的糖基[26],重新得到活化。正是这种糖基化修饰与脱糖基化平衡机制的存在,为抗生素生产菌提供了自我保护,而又不会使合成的抗生素永久性失活。
3 改造糖基的研究进展
一些糖基与抗生素的活性密切相关,可以通过基因工程对糖基进行改造,合成新结构的抗生素,来解决当前日益严重的致病细菌耐药性问题。其方法归纳起来有三种:①改变脱氧糖基结构;②替换连接的糖基;③去除糖基。
3.1 通过基因工程改变脱氧糖基结构
随着对抗生素上连接的糖基特别是脱氧糖基合成过程和具有灵活性的糖基转移酶的了解,可以通过两种方法来改变脱氧糖基结构,一是通过表达外源基因对脱氧糖基做进一步的修饰。1999年Zhao等[27]通过对红霉糖胺进一步的氨基化修饰合成了新结构红霉素衍生物。Gaisser[28]将来源于S.spinosa的甲基化酶基因spnI和spnK转入糖多孢红霉菌突变体SGT2中时,可把3?O?鼠李糖?红霉内酯B转化成3?O?(2"?O?甲基鼠李糖)?红霉内酯B和3?O?(2",3"?双?O?甲基鼠李糖)?红霉内酯B。如在糖多孢红霉菌SGT2中再转入eryCIII,则可以合成3?O?(2",3"?双?O?甲基鼠李糖)?红霉素D。二是失活糖基的某些合成基因。酒霉素上连接有红霉糖胺,敲除其相关合成基因desI、desII,利用糖基转移酶DesVII具有底物灵活性的特点,合成了一些红霉糖胺结构改变的酒霉素类似物[29,30]。红霉素含有mycarose和红霉糖胺两种脱氧糖,它们的合成和转移分别由6个eryB和6个eryC基因控制。通过破坏基因eryBII、eryBIII、eryBIV、eryBV和eryBVII,合成了mycarose结构改变的红霉素类似物[31,32];而破坏eryCIV则获得了带羟基红霉糖胺的红霉素A、B和C的衍生物[31]。
3.2 通过基因工程替换连接的糖基
Doumith[33]在研究基因oleP1、oleG1和oleG2生物功能时,发现oleG2编码的糖基转移酶OleG2可以将鼠李糖连接到6?dEB或EB的C?3位,合成C?3位为鼠李糖的新结构红霉素衍生物。2000年Aguirrezabalaga等[34]也在鉴定L?夹竹桃糖合成基因的时候合成了3?L?olivosyl?红霉内酯B等具有生物活性的抗生素。随后Gaisser[35]不仅证实了OleG2是个具有底物灵活性糖基转移酶,而且发现将来源于弗氏链霉菌的tylM2整合于糖多孢红霉菌突变体染色体上时,可以将外加的16?元环泰乐酮进行红霉糖胺糖基修饰。2004年Ward等[36]利用TylM2具有底物灵活性的特点,将chalcomycin的合成基因表达于突变的弗氏链霉菌株中,合成了将C?5位D?查耳糖(D?chalcose)替代为碳霉糖且C?3位为羰基16元环的类似物。
2001年Tang等[37]利用红霉糖胺转移酶DesVII具有底物灵活性的特点,将来源于委内瑞拉链霉菌的红霉糖胺糖合成酶和糖基转移酶基因整合到淡青紫链霉菌K4?114染色体上,构建了淡青紫链霉菌K39?22菌株,该菌株将添加到培养基中的narbonolide、10?deoxymethylnolide、6?dEB、3?keto?6?dEB进行红霉糖胺糖修饰,合成了具有生物活性的物质。2004年Hong等[38]在委瑞内拉链霉菌突变株内,利用具有底物灵活性的糖基转移酶DesVII,将来源于竹桃霉素合成菌株的TDP?4?keto?6?deoxy?D?glucose和urdamycin合成菌株的TDP?D?olivose添加到外加的12?和14?元环的内酯环上,合成了异鼠李糖(quinovose)或olivose糖基化的10?去氧微甘菊内酯(10?deoxymethynolide)和narbonolide活性物质。2006年Jung等[39]将泰乐酮合成基因tylG表达于突变的委瑞内拉链霉菌突变株内,在糖基转移酶DesVII作用下,合成了红霉糖胺糖基化的泰乐酮。
3.3 通过基因工程去除糖基
原来认为红霉素C?3位克拉定糖(cladinose)是抗菌活性的必需基团,C?3脱糖即意味着活性下降。1977年,Allen[40]证明了C?3位克拉定糖并不是红霉素抗菌活性所必须的基团,它的存在可诱导致病菌产生MLSB抗性。通过化学方法把C?3位克拉定糖水解,氧化为羰基,再进一步修饰所得到的以泰利霉素(telithromycin)为代表的酮内酯类抗生素依然保持着良好的抗菌活性,且对多种耐药菌有效。这为通过基因工程方法敲除糖基合成新结构的抗生素提供了可借鉴的途径。Xue[41]和McDaniel[42]在链霉菌体内合成了C?3位为羰基的6?dEB类似物,张部昌[43]、李凌凌[44]和曹孟婵[45]利用染色体同源重组在糖多孢红霉菌体内合成了C?3位为羰基的红霉内酯B类似物。
4 结束语
大环内酯类抗生素后修饰糖基的生物学功能研究已经取得了很大进展,了解糖基合成途径、糖基转移酶的结构特点和糖基生物学功能,对提高新结构抗生素产量,为非抗生素生产菌提供一种自我保护,使其合成抗生素的时候不至于产生自身中毒及改造糖基来合成新结构的抗生素等都具有重要的意义。
在改造糖基合成新结构抗生素方面的研究虽然也取得很大进展,但目前所有这些研究合成的抗生素产量都较低,绝大多数基因工程类似物都较原来的抗生素活性低。从现有的情况来看,改造糖基合成新的抗生素基因工程研究有三方面工作需要加强。一是提高产量,基因工程产物产量是衡量基因工程产品经济价值的一个重要指标,然而经过基因改造以后合成的类似物或其前体产量都普遍降低[15]。产量减少的原因可能是:基因重组蛋白结构不稳定;重组蛋白结构处于非最佳状态;对非天然底物利用效率低等,但产量降低的真正原因只有在研究聚酮合成酶空间结构及与底物的相互关系后才可能搞清楚[46]。二是加强高活性结构的筛选,加强基因工程类似物结构与活性关系的研究,以指导抗生素基因工程的定向改造。三是提高糖基转移酶底物灵活度,合成的抗生素或前体产量和活性在一定的程度上取决于糖基转移酶灵活度,可以通过突变等方法对糖基转移酶进行改造[15],为通过基因工程改造糖基合成新结构的抗生素提供强有力的工具。
【参考文献】
[1] 张部昌. 新酮内酯类抗生素组合生物合成的研究[D]. 北京:军事医学科学院生物工程研究所,2002
[2] 张部昌,赵志虎,马清钧. 红霉素生物合成的分子生物学[J]. 生物技术通讯,2001,12(2):151
[3] 张部昌,赵志虎,马清钧. 红霉素基因工程研究进展[J]. 中国生物工程杂志,2002,22(3):40
[4] Weissman K J. Polyketelide biosynthesis: understanding and exploiting modularity [J]. Phlos Transact A Math Phys Eng Sci,2004,362(1825):2671
[5] Trefzer A, Salas J A, Bechthold A. Genes and enzymes involved in deoxysugar biosynthesis in bacteria [J]. Nat Prod Rep,1999,16(3):283
[6] Piepersberg W. Pathway engineering in secondary metabolite?producing actinomycetes [J]. Crit Rev Biotechnol,1994,14(3):251
[7] Amann S, Drager G, Rupprath C, et al. (Chemo) enzymatic synthesis of dTDP?activated 2,6?dideoxysugars as building blocks of polyketide antibiotics [J]. Carbohydr Res,2001,335(1):23
[8] Melancon C E, Yu W L, Liu H W. TDP?mycaminose biosynthetic pathway revised and conversion of desosa?mine pathway to mycaminose pathway with one gene [J]. J Am Chem Soc,2005,127(35):12240
[9] Salah?Bey K, Doumith M, Michel J M, et al. Targeted gene inactivation for the elucidation of deoxysugar biosynthesis in the erythromycin producer Saccharopolyspora erythraea [J]. Mol Gen Genet,1998,257(5):542
[10] Xue Y, Zhao L, Liu H W, et al. A gene cluster for macrolide antibiotic biosynthesis in Streptomyces venezuelae: architecture of metabolic diversity [J]. Proc Natl Acad Sci USA,1998,95(21):12111
[11] Gaisser S, Reather J, Wirtz G, et al. A defined system for hybrid macrolide biosynthesis in Saccharopolyspora erythraea [J]. Mol Microbiol,2000,36(2):391
[12] Melancon C E, Takahashi H, Liu H W. Characterization of tylM3/tylM2 and mydC/mycB pairs required for efficient glycosyltransfer in macrolide antibiotic biosynthesis [J]. J Am Chem Soc,2004,126(51):16726
[13] Lee H Y, Chung H S, Hang C, et al. Reconstitution and characterizationof a new desosaminyl transferase EryCIII from the erythromycin biosynthetic pathway [J]. J Am Chem Soc,2004,126(32):9924
[14] Mulichak A M, Losey H C, Walsh C T, et al. Structure of the UDP?glucosyltransferase GtfB that modifies the heptapeptideaglycone in the biosynthesis of vancomycin group antibiotics [J]. Structure,2001,9(7):547
[15] Floss H G. Antibiotic biosynthesis: from natural to unnatural compounds [J]. J Ind Microbiol Biotechnol,2001,27(3):183
[16] Schlunzen F, Zarivach R, Harms J, et al. Structural basis for the interaction of antibiotics with the peptidyltransferase center in eubacteria [J]. Nature,2001,413(6858):814
[17] Fish S A, Cundliffe E. Structure?activity studies of tylosin?related macrolides [J]. J Antibiot,1996,49(10):1044
[18] Poulsen S M, Kofoed C, Vester B. Inhibition of the ribosomal peptidyl transferase reaction by the mycarose moiety of the antibiotics carbomycin, spiramycin and tylosin [J]. J Mol Biol,2000,304(3):471
[19] Rodriguez?Fonseca C, Phan H, Long K S, et al. Puromycin?rRNA interaction sites at the peptidyl transferase center [J]. RNA,2000,6(5):744
[20] Fish S A, Cundliffe E. Stimulation of polyketideme?tabolism in Streptomyces fradiae by tylosin and its glycosylated precursors [J]. Microbiology,1997,143(12):3871
[21] Butler A R, Flint S A, Cundliffe E. Feedback control of polyketide metabolism during tylosin production [J]. Microbiology,2001,147(4):795
[22] Waldron C, Matsushima P, Rosteck P R, et al. Cloning and analysis of the spinosad biosynthetic gene cluster of Saccharopolyspora spinosa [J]. Chem Biol,2001,8(5):487
[23] Jenkins G, Cundliffe E. Cloning and characterization of two genes from Streptomyces lividans that confer inducible resistance to lincomycin and macrolide antibiotics [J]. Gene,1991,108(1):55
[24] Zhao L, Beyer N J, Borisova S A, et al. Beta?glucosylation as a part of self?resistance mechanism in methymy?cin/pikromycin producing strain Streptomyces venezuele [J]. Biochemistry,2003,42(50):14794
[25] Sasaki J, Mizoue K, Morimoto S, et al. Microbial glycosylation of macrolide antibiotics by Streptomyces hygroscopicus ATCC 31080 and distribution of a macrolide glycosyltransferase in several Streptomyces strains [J]. J Antibiot (Tokyo),1996,49(11):1110
[26] Vilches C, Hernandez C, Mendez C, et al. Role of glycosylation and deglycosylation in biosynthesis of and resistance to oleandomycin in the producer organism, Streptomyces antibioticus [J]. J Bacteriol,1992,174(1):161
[27] Zhao L, Ahlert J, Xue Y, et al. Engineering a methy?mycin/pikromycin?calicheamicin hybrid: construciton of two new macrolides carrying a designed sugar moiety [J]. J Am Chem Soc,1999,121(6):9881
[28] Gaisser S, Lill R, Wirtz G, et al. New erythromycin derivatives from Saccharopolyspora erythraea using sugar O?methyltransferases from the spinosyn biosynthetic gene cluster [J]. Mol Microbiol,2001,41(5):1223
[29] Borisova S A, Zhao L, Sheman D H, et al. Biosynthesis of desosamine: construction of a new macrolide carrying a genetically designed sugar moiety [J]. Org Lett,1999,15(1):133
[30] Zhao L, Borisova S A, Yeung S M, et al. Study of C?4 deoxygenation in the biosynthesis of desosamine: evidence implicating a novel mechanism [J]. J Am Chem Soc,2001,123(32):7909
[31] Salah?Bey K, Doumith M, Michel J M, et al. Targeted gene inactivation for the elucidation of deoxysugar biosynthesis in the erythromycin producer Saccharopo?lyspora erythraea [J]. Mol Gen Genet,1998,275(5):542
[32] Summers R G, Donadio S, Staver M J, et al. Sequencing and mutagenesis of genes from the erythromycin biosynthetic gene cluster of Saccharopolyspora erythraea that are involved in L?mycarose and D?desosamine production [J]. Microbiology,1997,143(10):3251
[33] Doumith M, Legrand R, Lang C, et al. Interspecies complementation in Saccharopolyspora erythraea: elucidation of the function of oleP1, oleG1 and oleG2 from the oleandomycin biosynthetic gene cluster of Streptomyces antibioticus and generation of new erythromycin derivatives [J]. Mol Microbiol,1999,34(5):1039
[34] AguirrezabalagaI, Olano C, Allende N, et al. Identification and expression of genes involved in biosynthesis of L?Oleandrose and its intermediate L?Olivose in the oleandomycin producer Streptomyces antibioticus [J]. Anti?microb Agents Chemother,2000,44(5):1266
[35] Gaisser S, Reather J, Wirtz G, et al. A defined system for hybrid macrolide biosynthesis in Saccharopolyspora erythraea [J]. Mol Microbiol,2000,36(2):391
[36] Ward S L, Hu Z, Schirmer A, et al. Chalcomycin biosynthesis gene cluster from Streptomyces bikiniensis: novel features of an unusual ketolide produced through expression of the chm polyketide synthase in Streptomyces fradiae [J]. Antimicrob Agents Chemother,2004,48(12):4703
[37] Tang L, McDaniel R. Construction of desosamine containing polyketide libraries using a glycosyltransferase with broad substrate specificity [J]. Chem Biol,2001,8(6):547
[38] Hong J S, Park S H, Choi C Y, et al. New olivosyl derivatives of methymycin/pikromycin from an engineered strain of Streptomyces venezuelae [J]. FEMS Microbiol Lett,2004,238(2):391
[39] Jung W S, Lee S K, Hong J S, et al. Heterologous expression of tylosin polyketide synthase and production of a hybrid bioactive macrolide in Streptomyces venezuelae [J]. Appl Microbiol Biotechnol,2006,72(4):763
[40] Allen N E. Mcrolide resistance in Staphylococcus aureus: inducers of macrolide resistance [J]. Antimicrob Agents Chemother,1977,11(4):669
[41] Xue Q, Ashley G, Hutchinson C R, et al. A multiplasmid approach to preparing large libraries of polyketides [J]. Proc Natl Acad Sci USA,1999,96(21):11740
[42] McDaniel R, Thamchaipenet A, Gustafsson C, et al. Multiple genetic modifications of the erythromycin polyketide synthase to produce a library of novel′unnatural′ natural products [J]. Proc Natl Acad Sci USA,1999,96(5):1846
[43] 张部昌,赵志虎,王以光,等. 合成酮内酯类3?脱氧?3?羰基?红霉内酯B糖多孢红霉菌M的构建[J]. 生物工程学报,2002,18(1):198
[44] 李凌凌,张部昌. 糖多孢红霉菌λ30?SRR突变体构建及产物鉴定[J]. 军事医学科学院院刊,2004,28(4):314
[45] 曹孟婵,张部昌,马清钧. 糖多孢红霉菌A226?YA突变体构建及产物分析[J]. 军事医学科学院院刊,2006,30(5):428
[46] Gokhale R S, Tsuji S Y, Cane D E, et al. Dissecting and exploiting intermodular communication in polyketide synthases [J]. Science,1999,284(5413):482